
11 minute read
Genoma, transpo sones y secuen cias Alu
Genoma, transposones y secuencias Alu
Por Raquel Buj
Advertisement
El término genoma fue acuñado por Hans Winkler en La Dra. McClintock se percató de que dentro de una mazorca convivían granos de varios colores, y que incluso las células de un organismo desarrollado a partir de un dor) cambiaba entre diferentes células de una misma maeste descubrimiento, la comunidad científica no le prestó mayor atención, sin embargo con el paso de los años fueron apareciendo más ejemplos en otras especies que apoyaban la idea de McClintock. El nombre “elementos controladores” fue sustituido por el de transposones o coloquialmente “genes saltarines” por su capacidad para copiarse e insertarse en nuevas posiciones genómicas [3, 4]. La Dra. McClintock fue galardonada con el Premio
1920 como “un baúl de viaje para genes y cromosomas”. Actualmente entendemos el genoma como la totalidad de la información genética presente en un organismo. Cada uno de nosotros tiene un genoma exclusivo que, sin embargo, es similar en un 99.8% al genoma del resto de las personas. Es esta diferencia del 0.2% la

diferentes zonas de un mismo grano podía presentar colores distintos. Esto contradecía las leyes de la herencia tal y como se conocían en aquel momento, ya que si el color es una característica heredada de los padres, todas único zigoto deberían tener el mismo color. Durante su investigación, McClintock descubrió que la posición cromosómica de dos secuencias génicas (Activador y Disociazorca. Estableció que en el maíz existían unos “elementos controladores” que podían mover su posición alterando el color de los granos de maíz. Cuando se hizo público que nos hace ser únicos en el mundo.
Nobel de Medicina y Fisiología en 1983 siendo la primera
La información que esconde el genoma humano fue descifrada en 2003 gracias a un consorcio público internacional (Proyecto Genoma Humano) y a la empresa privada Celera. A partir del análisis de esta información sabemos que únicamente el 1.5% del genoma son genes, el resto corresponde al llamado DNA no codificante (35.5%) y DNA repetitivo (63%). Este último Barbara McClintock (1902-1992). Sello expedido por el Servicio Postal de los hace referencia a varias secuencias EE.UU. 2005. de DNA que se encuentran en múltiples copias dentro del genoma. Podemos diferenciar dos mujer en recibir este galardón a título personal en dicha tipos: DNA en tándem (microsatélites, minisatélites y tecategoría. lómeros) y DNA disperso, dentro del que se engloban los “elementos móviles” o transponibles del genoma (SINEs, Desde entonces y gracias a la mejora en las técnicas de seLINEs, LTR y transposones) [1, 2]. cuenciación, sabemos que todos los genomas eucariotas estudiados contienen elementos transponibles, aunque ¿Qué es un elemento transponible? hay una gran variedad en cuanto a proporción y actividad. De hecho, muchos investigadores consideran a estos Los elementos transponibles son secuencias de DNA que elementos como una plaga intracelular que ataca a los puede moverse de forma autosuficiente por el genoma genomas hospedadores explotando sus recursos celulaen un proceso denominado transposición. Este fenómeno res [1]. Tanto es así que, en algunos casos esta plaga es fue descubierto en la década de los 40 por la estadounicapaz de explicar por sí sola la paradoja del valor-C. Este dense Bárbara McClintock mientras estudiaba la citogeenigma viene a cuestionar el por qué organismos sencillos
tienen más cantidad de DNA que otros más complejos. Por poner un ejemplo, Hordeum vulgare (cebada cultivada) tiene un valor-C de 5Gb y un porcentaje de elementos transponibles del 55% [5, 6].
Al ser estas secuencias una plaga, cabría esperar que la selección negativa acabase con ellas, sin embargo no lo hace. Hay dos teorías al respecto, una propone que la selección purificadora (negativa) no es tan fuerte como para impedir la acumulación de estos elementos. La otra argumenta que esta plaga se tolera debido a su ocasional influencia positiva sobre la evolución de los genomas. Es esta última hipótesis la que ha cobrado mayor importancia en los últimos años [7, 8]. De todos los elementos móviles que se encuentran en el genoma humano, las secuencias Alu son las más abundantes. Se han contado 1.1 millones de estas secuencias en el genoma, esto es un Alu cada 4 Kb lo que supone un 10% de la masa total del genoma [1, 9]. Box A y B en la figura 1) [11]. El movimiento de estas secuencias (retrotransposición) ocurre en tres pasos [Figura 2]. En primer lugar la RNA polimerasa III de la célula hospedadora transcribe la secuencia Alu a un RNA a partir del promotor interno (Box A y B). Como dicha secuencia no tiene señal de terminación, el transcrito se extiende hasta que aparece alguna. A continuación, una endonucleasa L1 corta su diana en algún lugar del genoma dejando un extremo rico en timinas al que se une el transcrito. Utilizando este extremo como primer, la transcriptasa inversa retrotranscribe el RNA a DNA mediándose así la ligación e integración de la Alu en un nuevo sitio genómico. Por su parte, la retrotransposición de la hebra complementaria continúa sin resolverse [1].
Las secuencias Alu, siguiendo el modelo vírico, se apoderan de la maquinaria celular para sus propios fines tomando prestadas las enzimas del hospedador. Sin embargo, los genomas eucariotas están desprovistos de transcriptasa inversa y endonucleasa L1, entonces ¿De dónde obtienen estas enzimas si como hemos mencionado anteriormente no posee ninguna ORF? Parece ser que en su expansión, las Alu se aprovechan de los productos de otros transposones del genoma humano como son los LINEs (Long Interspersed Nuclear Elements) que sí codifican la transcriptasa inversa y la endonucleasa L1 [6, 12].
¿Cuál es el origen las secuencias Alu?


¿Qué es una secuencia Alu? Los estudios filogenéticos basados en las secuencias Alu Las secuencias Alu son un tipo de retrotransposones determinan que su origen y amplificación tuvo lugar hace (transposones de RNA) exclusivos de los genomas de 65 millones de años, coincidiendo con la expansión de primates. Clasificadas como SINEs (Short Interspersed los primates. Provienen del 7SL-RNA, componente riboNucleolar Elements) reciben su nombre de la diana de nucleico esencial de la SRP (Señal de Reconocimiento de restricción AluI que poseen. Su distribución dentro del Partículas) encargada de reconocer específicamente el genoma humano no es aleatoria, si no que tienden a acupéptido señal de las proteínas que han de ser translocamularse preferiblemente en regiones ricas en genes [1, das al retículo endoplasmático [9]. 10]. En un primer momento el 7SL-RNA sufrió una deleción Tienen un tamaño de 300 nucleótidos y están formadas de su parte central a la que se le unió un oligo-d(A) forpor dos monómeros similares colocados en tándem y mando el monómero Alu fósil (FAM). A continuación se separadas por una secuencia oligo-d(A) (un continuo de partió en dos dando lugar al monómero Alu fósil izquierdo nucleótidos adenina de número variable). Carecen de (FLAM) y al monómero Alu fósil derecho (FRAM). Ambos pauta abierta de monómeros evolulectura (ORF), es cionaron por sedecir, no contieparado hasta que nen ninguna sehace aproximadacuencia que codimente 65 millones fique gen alguno. de años volvieron a No obstante, el encontrarse y a dimonómero demerizar, originando recho presenta la primera secuenuna región rica cia Alu. El proceso en adeninas en por el cual estos su extremo 3’ y el dos monómeros se izquierdo el proreencontraron tomotor de la RNA davía no ha sido repolimerasa III (resuelto [9, 1, 13]. presentado como Figura 1. Estructura de una secuencia Alu. Actualmente sólo
polimerasa III y la transcriptasa inversa. un pequeño porcentaje de las Alu, conocidas siguiendo el proceso de la retrotransposición. El resto han perdido esta capacidad debido a la acumulación gradual de mutaciones [13]. Sin embargo, la secuencia 7SL-RNA está más o menos conservada en todas las especies de primates, lo que sugiere la presencia de FLAM y FRAM en todos los linajes [9]. Las Alus se han insertado en los genomas de primates con tasas variables, la más alta se postula que tuvo lugar hace 55 millones de años generando la subfamilia Alu-Sx. En los últimos 4 a 6 millones de años, después de la divergencia entre humanos y monos africanos, se dieron algunas expansiones de subfamilias de Alus “jóvenes” que todavía no se han fijado en el genoma humano. Esto genera un polimorfismo debido a la presencia y/o ausencia de dichas secuencias [Figuras 3 y 4] [1, 9]. La expansión de estas secuencias en el genoma tiene varias consecuencias resultado de su inserción, expansión y tipo de secuencia.

La retrotransposición de una Alu a una nueva posición genómica puede ser evolutivamente favorable. Muchas subfamilias de Alus tienen sitios de unión a proteínas que eventualmente pueden actuar como represores o potenciadores de la expresión génica. Si una de estas se inserta en las secuencias reguladoras de la expresión de un gen, podría otorgarle nuevas funciones que en ocasiones pueden ser evolutivamente favorables. En la tabla 1 se exponen algunos ejemplos en los que las inserciones han contribuido a modifica positivamente la expresión de
Figura 2. Retrotransposición de una secuencia Alu mediada por la RNA

como master, son susceptibles de expandirse

¿Cómo afecta al genoma la presencia de las secuencias Alu?
algunos genes [14].

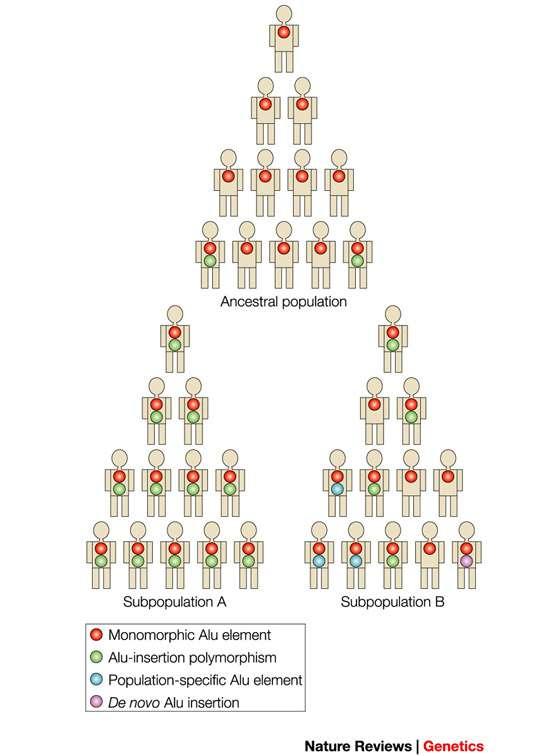
Sin embargo esto no es lo normal, generalmente la translocación de secuencias en las regiones reguladoras es muy perjudicial, ya que distorsiona el patrón de expresión de un gen bien regulado. Por otro lado, la inserción de una Alu dentro de la secuencia codificante de un gen, provoca la ruptura de su pauta de lectura. Esto hace que el gen sea incapaz de codificar una proteína correcta, lo que tiene evidentes efectos negativos. De hecho algunas enfermedades como neurofibromatosis, cáncer de mama o Sindrome de Apert están asociadas a este tipo de inserciones.
Por otro lado y de forma natural, durante la meiosis que origina las células germinales, tiene lugar la llamada recombinación homóloga. Este proceso consiste en el intercambio de información entre los cromosomas homólogos, es decir, entre cada uno del par de cromosomas que existe dentro del organismo eucariota diploide. Esto promueve la diversidad genética y explica el por qué los hermanos (hijos de los mismos padres) pueden ser radicalmente diferentes. Todos contienen la mitad del DNA de su padre y la mitad de su madre, pero estas mitades no tienen por qué ser necesariamente las mismas gracias a la recombinación homóloga.
Cuando esta recombinación tiene lugar entre dos secuencias de un par de cromosomas no homólogos se dice que es desigual o no homóloga y causa deleciones, duplicaciones y translocaciones de material genético. Debido a la elevada homología de secuencia entre las Alus y a su alta frecuencia en el genoma, es habitual que se produzcan este tipo de recombinaciones con el consecuente efecto negativo sobre el genoma. Algunos de los desórdenes asociados son la enfermedad de Tay-Sachs, α-talasemia, el síndrome de Lesch-Nyhan o la hipercolesterolemia familiar. Con todo, en algunos momentos este proceso ha sido beneficioso generando diversidad, un ejemplo es la diversificación de los genes de la tropoelastina durante la evolución de los primates [1].

En conclusión, el estudio del origen de las secuencias Alu, así como su mecanismo de expansión y diversificación, nos ayuda a entender un poquito mejor la evolución de los genomas. Como hemos visto, los polimorfismos causados por la inserción de Alus son buenos marcadores en los estudios de evolución, ya que se producen por un único evento mutacional de manera que podemos conocer el estado actual y ancestral (de no inserción). Además, la expansión de las secuencias Alu causa múltiples patologías. Se estima que aproximadamente el 0.5% de los desórdenes genéticos en humanos se deben directamente a la inserción o recombinación no homóloga de las Alus.
3. The significance of responses of the genome. McClintock B. Science. 1984. 226:792-801.
4.
5.
6.
7.
8. Two genes, no enzyme: a second look at Barbara McClintock and the 1951 Cold Spring Harbor Symposium. Comfort NC. 1995. 140(4):1161-6.
The influence of transposable elements on genome size. Biémont C, Vieira C. J Soc Biol. 2004. 198(4):413-7.
Reverse transcriptase encoded by a human transposable element. Mathias SL,Scott AF,Kazazian HH Jr,Boeke JD, Gabriel A. Science. 1991. 20;254(5039):1808-10.
Selfish DNA: the ultimate parasite. Orgel LE, Crick FH. Nature. 1980. 17;284(5757):604- Genomic scrap yard: how genomes utilize all that junk. Makałowski W. Gene. 2000. 23:259(1.2):61-7.
9. Alu sequences. Mighell AJ, Markham AF, Robinson PA. FEBS Lett. 1997. 3;417(1):1-5.
10. Alu distribution and mutation types of cancer genes. Zhang W, Edwards A, Fan W, Deininger P, Zhang K. BMC Genomics. 2011. 23;12:157.
11. Alu elements as regulators of gene expression. Häsler J, Strub K. Nucleic Acids Res. 2006;34(19):5491-7.
12. LINE-mediated retrotransposition ofmarked Alu sequences. Dewannieux M, Esnault C, Heidmann T. Nat Genet. 2003. 35(1):41-8.
13. Evolutionary history of 7SL RNA-derived SINEs in Supraprimates. Kriegs JO, Churakov G, Jurka J, Brosius
J, Schmitz J. Trends Genet. 2007. 23(4):158-61.
14. DNA sequence insertion and evolutionary variation in gene regulation. Britten RJ. Proc Natl Acad Sci U S A. 1996. 3;93(18):9374-7.
Raquel Buj es una estudiante de doctorado amante de los gatos, y cuando no está entre pipetas podéis encontrarla en su blog ‘Locos por la Biología’ o en Twitter, como @Bioyupi
Bibliografía:
1.
2. Alu repeats and human genomic diversity. Batzer MA, Deininger PL. Nat Rev Genet. 2002. 3(5): 370-9.
A unified classification system for eukaryotic transposable elements. Wicker T, Sabot F, Hua-Van A, Bennet-Wicker T, Sabot F, Hua-Van A, Bennetzen JL, Capy P, Chalhoub B, Flavell A, Leroy P, Morgante







