

Your Agilent Cell Imaging Just Got Brainy
New Neurite Outgrowth Analysis for Agilent BioTek Imagers

Agilent BioTek Gen5 Neurite Outgrowth Module for Agilent BioTek Cytation and Lionhear t Imagers
The new Agilent BioTek Gen5 neurite outgrowth module delivers the ability to analyze neuronal cell dynamics from fluorescently-labeled or label-free cell images captured in end point or kinetic workflows.
New Oppor tunities for Neuroscience Researchers
The Gen5 neurite outgrowth module suppor ts research in neuropathology, developmental biology, neurotoxicity screening, and regenerative medicine. The module is compatible with Gen5 Image Prime software for use with BioTek Lionhear t automated microscopes, Cytation C10 confocal imaging reader, and Cytation cell imaging multimode readers.
Learn more at https://aglt.co/maFH

Key Metrics Provided by the Gen5 Neruite Outgrowth Module
• Soma count
• Total neurite length (μm)
• Average neurite length (μm)
• Total neurite branches
• Average neurite branches
• Total neurite area (μm2)
• Neurite thickness (μm)
• Neurite count
• Average neurite count
Agilent BioTek Cytation C10 confocal imaging reader
Agilent BioTek Cytation 7 cell imaging multimode reader
Agilent BioTek Lionhear t FX automated microscope
Neurite images are skeletonized to calculate impor tant metrics.

IMAGEN PORTADA
Número 222
DICIEMBRE 2024
SEBBM es una publicación periódica de la Sociedad Española de Bioquímica y Biología Molecular © SEBBM. Los artículos y colaboraciones reflejan la opinión de sus autores y no necesariamente la opinión de la SEBBM. Se autoriza la reproducción del contenido, siempre que se cite la procedencia.
Sociedad Española de Bioquímica y Biología Molecular C/ Ramiro de Maeztu, 9 28040 Madrid
Telf.: +34 681 916 770 e-mail: sebbm@sebbm.es http://www.sebbm.es
Directora: Inmaculada Yruela Guerrero
Director honorario: Joan J. Guinovart
Directora adjunta: Ana Mª Mata
Consejo editorial: Antonio Ferrer Montiel, Isabel Varela Nieto, Vicente Rubio, Federico MayorMenéndez, Miguel Ángel de la Rosa.
SECCIONES:
Entrevista y Política
científica: Ismael Gaona Pérez
Investigación: Joaquim Ros
Educación universitaria: Ángel
Herráez
Reseñas de libros: Juli Peretó
Sociedad: Carmen Aragón
Empresas: María Monsalve
Coordinación del número 222: Ana María Rojas Mendoza
Diseño: Filoestudio
Depósito legal: M-13490-2016
ISSN: 1696-4837
TRIBUNA
El próximo cuatrienio
Antonio Ferrer
EDITORIAL
Nueva etapa de la mano de la IA
Inmaculada Yruela Guerrero
DOSIER CIENTÍFICO
La Inteligencia artificial en Biociencias
Ana María Rojas Mendoza
La Inteligencia artificial como motor de la medicina de precisión: predecir y explicar para simular
Lucía Sánchez García, Inés Rivero, Fátima Sánchez-Cabo
La revolución de la Inteligencia artificial en la biología estructural de proteínas
Pablo Chacón Montes
Inteligencia Artificial Generativa (GenAI) para diseñar genomas de organismos digitales
Francisco J. Borrallo-Vázquez y Miguel A. Fortuna
Anotación de función en proteínas usando modelos de lenguaje
Ana M. Rojas Mendoza, Ildefonso Cases, Gemma MartinezRedondo, Rosa Fernández
ENTREVISTA
Izaskun Lacunza. Directora general de la FECYT
Ismael Gaona
POLÍTICA CIENTÍFICA
Vocaciones: la crisis silenciosa
Ismael Gaona
A FONDO
Los Premios Nobel de Física y de Química de 2024
Javier Sancho
EDUCACIÓN UNIVERSITARIA
¿Podemos mejorar nuestros pósteres? ¡Sí, podemos!
Carlos Gancedo
Resumen y consideraciones finales extraídas del simposio de educación en el marco del 46º congreso de la SEBBM
Marina Lasa
INVESTIGACIÓN
Joaquim Ros
IN MEMORIAM
Antonio Sillero (1938-2024)
Carlos Gancedo
Claudio Fernández de Heredia
SOCIEDAD
Noticias de actualidad de la SEBBM
RESEÑA DE LIBROS
Hay biología (y mucha) más allá del ADN
Víctor de Lorenzo
PUBLIRREPORTAJES
Agilent
Controltécnica
Condalab
Ecogen Kühner
Aplitech
Proquinorte

Antonio Ferrer
Presidente SEBBM
El próximo cuatrienio...
Comienzo mi primera tribuna desde la presidencia de la SEBBM con el empeño de asentar la labor realizada por Isabel Varela que ha promovido una plétora de acciones en la gestión, así como en la visibilidad nacional e internacional de la SEBBM, y en su influencia social. Por destacar algunos logros, el primero sería la modernización de la web, complementada este año con la publicación del blog, una herramienta de divulgación de los socios y para los socios y al que todos debiéramos contribuir. Otra acción destacable ha sido el establecimiento de acuerdos con las Reales Academias Nacionales de Farmacia (RANF), Ciencias Naturales y Exactas (RAC) y de Medicina (RANM) para la realización de conferencias y actos conjuntos. En 2024, hemos disfrutado de las conferencias impartidas por Vicente Rubio en la RANF y de Isabel Fariñas en la RAC, así como de la semana Severo Ochoa en la RANM, co-organizada por la Fundación Carmen y Severo Ochoa (FCySO) y la Universidad de Nebrija, con las conferencias de Vicente Rubio, Federico Mayor y María Blasco. Además, y también junto con la FCySO se realizó la 2ª Conferencia Severo Ochoa en Bilbao coordinada por María Ángeles Serrano, y organizada por Rosa Barrio, Alicia Alonso y Nerea Osinalde. Por último, quiero destacar la excelencia del 46º Congreso SEBBM organizado por María Mayán en Coruña. Hemos disfrutado de una agenda intensa e interesantísima fruto de la amplia labor de la anterior junta directiva. No menos importante ha sido la excelente gestión en la tesorería que ha potenciado la salud económica de la SEBBM que permitirá abordar, con cierta tranquilidad, los objetivos y acciones del próximo cuatrienio.
En la nueva junta, que tomó posesión el pasado septiembre, me acompañan Irene Díaz Moreno como vicepresidenta, Lluís Montoliu como secretario, María Monsalve como tesorera, Lourdes Ruiz como secretaria-electa, y en las vocalías, Guadalupe Sabio en cónsules y mujer y ciencia, Raúl Estévez en mentoría y comunicación, Sara García en jóvenes y educación, Patricia González en congresos y talleres, Patricia Aspichueta en grupos, y Marina García en socios protectores y
patrocinadores. Además, como co-optados tenemos a Isabel Varela (expresidenta), Inmaculada Yruela (directora de la Revista SEBBM), y Marina Tapias (coordinadora de la sección junior). Marina García sustituye a María Ángeles Almeida que ha sido nombrada coordinadora de programas del ISCIII y a la que le damos todo nuestro apoyo para una gestión exitosa.
La nueva junta entra con la energía y la ilusión de impulsar en el próximo cuatrienio un incremento del número de socios en todas sus categorías, la consolidación y la expansión de las relaciones Institucionales e Internacionales, y el fortalecimiento e internacionalización de los congresos SEBBM. Además, también nos marcamos como meta promover una participación más activa de los jóvenes en los congresos a través del apoyo a las iniciativas de la sección junior, fortalecer las acciones y reuniones de los grupos científicos, consolidar la conferencia Severo Ochoa para la excelencia científica, facilitar la formación transversal de nuestros jóvenes investigadores, y fomentar estrategias y herramientas de apoyo para la transferencia y/o traslación de los resultados de los socios SEBBM.
Un logro por destacar, y para el que vamos a trabajar intensamente los próximos años, es la organización del Congreso FEBS-IUBMB-SEBBM en el año 2030 al haber sido mayoritariamente apoyada la propuesta presentada por Isabel Varela e Irene Díaz en el congreso IUBMB 2024 en Melbourne. Cabe recordar que este congreso lo organizó la SEBBM en Sevilla en 2012 bajo la presidencia de Miguel Ángel de la Rosa y fue todo un éxito en la proyección y reconocimiento internacional de nuestra sociedad. Confiamos en cosechar un éxito similar en 2030. En cuanto a la sede, existen dos candidaturas Madrid y Sevilla cuya decisión final está condicionada a una auditoria de las infraestructuras locales por parte de IUBMB y FEBS en 2025.
Una de las primeras acciones que ha tomado la nueva junta directiva ha sido promover la constitución de la Fundación SEBBM (FSEBBM) con el objetivo de acercar la SEBBM a la sociedad, impulsando proyectos de mayor ámbito social, tanto a nivel de divulgación social de la ciencia como de formación e información del público en general. Además, la creación de una Fundación facilita la canalización y gestión de donaciones de nuestros socios, de entidades privadas y, de la gente en general. Esta iniciativa ha surgido debido a que la SEBBM no es reconocida como una asociación de interés público lo que limita su capacidad para obtener donaciones fiscalmente deducibles. Los objetivos fundacionales establecidos son: (i) favorecer el contacto y la interacción científica en el ámbito de la bioquímica y la biología molecular, así como una interacción interdisciplinar; (ii) potenciar la investigación y mejorar la docencia y la cultura científica en el campo de las biociencias; e, (iii) impulsar la divulgación más amplia y profunda sobre aquellos aspectos sociales y científicos de las biociencias a la sociedad. Para alcanzar estos objetivos se establecerán diversas acciones que permitirán acercar el progreso en el ámbito de la bioquímica y la biología molecular y, en general, de las biociencias al público. A su vez, se establecerán programas para fomentar el interés de los jóvenes por esta rama del conocimiento.
Iniciamos esta etapa con la intención de impulsar el crecimiento de la SEBBM. Una acción esencial para alcanzar este objetivo es nuestro congreso anual. Quiero recordar que en 2025 el congreso será en Cáceres del 2 al 5 de septiembre bajo la presidencia de Guadalupe Sabio, en una magnífica sede como es el Complejo Cultural San Francisco y la ciudad de Cáceres, y con un programa de excelencia que se está elaborando gracias a las propuestas de simposios que ha recibido el comité organizador. Además, siendo conscientes de la importancia de las reuniones de grupo, vamos a priorizar que todos los grupos tengan su espacio y tiempo durante el congreso. ¡Os esperamos en Cáceres!

Inmaculada
Yruela Guerrero
Directora Revista SEBBM
Nueva etapa de la mano de la IA
Con el número de diciembre de 2024 inicio, con ilusión, mi etapa como directora de la Revista SEBBM. Agradezco a Antonio Ferrer, ahora presidente de la Sociedad, a Ana Mª Mata, directora adjunta y al comité editorial, la confianza que han depositado en mí para continuar esta empresa. El camino, primero emprendido y luego continuado con empeño, ingenio y maestría, por los que me precedieron -Carlos Gancedo, Joan Guinovart, Miguel Ángel de la Rosa, Antonio Ferrer-, invita a proseguirlo con renovado empeño. También agradezco el apoyo y colaboración de los responsables de las diferentes secciones de la revista que continúan al frente: Carmen Aragón, Ángel Herráez, María Monsalve, Juli Peretó, Joaquim Ros, Ismael Gaona.
Tal como mostraba la exposición Revista SEBBM: un observatorio de la actividad y política científicas en España, presentada el pasado año con motivo del sexagésimo aniversario de la SEBBM en el Congreso de Zaragoza, la revista ha tenido diferentes etapas desde que empezó a rodar en 1963 –circulares, boletines, revista-. Cada una de ellas vino acompañada de un cambio en el diseño y en los contenidos. Hoy, la renovada Revista SEBBM también incorpora novedades –cambio en el diseño de la portada, rotulación, maquetación y publicación-. A partir de ahora la revista se publica sólo en formato digital, accesible a través de nuestra web https://sebbm.es/revista/. Terminamos el año conociendo los galardonados con los Premios Nobel en Fisiología o Medicina, en Física y en Química. Tres premios que reconocen investigaciones de gran impacto científico y social, muy ligadas a la actividad que realizan grupos de socias y socios SEBBM. Y dos de ellos especialmente conectados con este número de diciembre, dedicado a la ‘Inteligencia artificial en Biociencias’ cuyo dosier ha coordinado Ana Mª Rojas, investigadora del CSIC en el Centro Andaluz de Biología del Desarrollo (CABD). Al encargar el dosier, por cierto, no se conocía el fallo de los premios, pero éramos muy conscientes de la actualidad e importancia del tema. Los premios Nobel 2024 en Física y en Química reconocen trabajos que sientan las bases para el
aprendizaje de las computadoras, necesarias para el desarrollo de la ‘Inteligencia artificial’ (IA), y que han conducido al diseño computacional de proteínas, y a la predicción de estructuras proteicas con su ayuda. Os invito a leer el análisis de Javier Sancho sobre este tema en la sección ‘A Fondo’.
Los trabajos de los galardonados han demandado grandes dosis de creatividad, ingenio y audacia, a la vez que profundos conocimientos básicos. Y también importantes dotes para la comunicación de los resultados y de los logros que han ido consiguiendo. Este es un aspecto fundamental para captar los recursos necesarios para la investigación y para atraer el interés de la comunidad científica. La comunicación de calidad es una herramienta esencial para aumentar el impacto de las investigaciones, establecer redes de colaboración y, en el caso del desarrollo de aplicaciones, conseguir fieles usuarios de las mismas. El artículo de Carlos Gancedo en la sección ‘Educación’ da un toque de atención sobre algunas cuestiones prácticas relacionadas con ello.
Todo esto me lleva a reflexionar sobre la importancia de la interdisciplinariedad, la comunicación y la divulgación en la actividad científica, dado que también estas tienen un aspecto social. La comunicación científica es cultura. El modo de relacionarnos ha cambiado vertiginosamente en la última década. No somos sólo individuos sociales, sino individuos socio-tecnológicos, estrechamente vinculados a las tecnologías digitales, de las que dependemos quizá demasiado. Ya se ha acuñado la expresión Homo digitalis. Nuestra realidad, actividad, comportamiento e interacciones se están transformando en información cuantificable (datos) que pueden ser medidos, analizados y explotados. La ‘Inteligencia artificial’ ha comenzado a alterar esa interdependencia. Los cambios nos están obligando a rediseñar las estrategias de comunicación y divulgación, y nos hacen reflexionar sobre si realmente nuestros mensajes llegan de forma fiable a los destinatarios finales y cumplen su objetivo.
¿Hay sobresaturación de canales y mensajes? Es patente que el trabajo científico y la transferencia de resultados se están viendo beneficiados de la era digital y de la IA. Pero también hay incertidumbres y cuestiones éticas que resolver. ¿Se ve afectada la autoría, la creatividad, la responsabilidad o la veracidad de los resultados y mensajes?
La proliferación de bulos y noticias falsas es una amenaza importante para la sociedad. También se cuestiona la neutralidad de los productos basados en IA y sus sesgos. Los gobiernos europeos no son ajenos a estas cuestiones. Para regular su uso, en abril de 2021, la Comisión Europea propuso el primer marco regulador de la UE para la IA, y el 12 de julio de 2024 se publicó la “Ley de Inteligencia Artificial” que entró en vigor el 1 de agosto. La Ley está diseñada para garantizar que el desarrollo de la IA y utilización en la UE sea fiable y proteja los derechos fundamentales de las personas.
La comunidad científica, así como las academias, fundaciones y sociedades científicas, incluida la SEBBM, realizan un gran esfuerzo para canalizar actividades fiables de difusión de la ciencia y del conocimiento científico actual que lleguen a todo tipo de público, tanto especializado como público general. Es todo un reto adaptarse a estos nuevos tiempos.
No puedo terminar esta editorial sin expresar mi dolor y tristeza por las consecuencias y personas fallecidas a causa de la DANA que tan trágicamente ha afectado a la Comunitat Valenciana, y a otras comunidades, los últimos días del pasado octubre. Mucho ánimo a los que os habéis visto afectados.
LA INTELIGENCIA ARTIFICIAL EN BIOCIENCIAS
Ana María Rojas Mendoza
Centro Andaluz de Biología del Desarrollo (CABD), CSIC, Sevilla. Grupo de Biología Computacional y Bioinformática. (Conexión BCB.Hub)
Biología e Inteligencia artificial (IA) han sido socios reticentes: cada uno va a lo suyo. Como en un mal baile, han interactuado sin coordinación y a ritmos diferentes. La Biología es sutil, subjetiva y cambiante, moviéndose en un contexto donde la excepción es quizá la regla. En contraste, la IA es una máquina insaciable que requiere grandes volúmenes de datos, homogéneos y estructurados. Con datos tan escasos y variables… ¡es difícil que la danza funcione!
Sin embargo, existen esos espacios de encuentro donde ambas pueden alinearse. En el campo de la biología computacional, de hecho, la IA ha estado presente desde hace décadas. La IA se ha empleado en tareas como la clasificación de secuencias biológicas (proteínas, ADN), destacando en la predicción de estructuras secundarias de proteínas. Desde su incepción en la década de los 80, C. Sander junto a B. Rost la re-implementó en redes neuronales para el mismo propósito. Desde entonces estas metodologías forman parte del arsenal metodológico de la bioinformática.
La IA incluye tanto las técnicas
tradicionales de aprendizaje automático (Machine Learning, ML) como las más avanzadas de aprendizaje profundo (Deep Learning, DL). La aplicación más común y recomendada de estas técnicas es en su modalidad “tradicional”, es decir, sin el uso de redes neuronales, para construir clasificadores supervisados (como la detección de “cáncer/no-cáncer”) o identificar patrones en conjuntos de datos no supervisados (como los genes sobre-expresados). Estas aplicaciones han tenido éxito y han contribuido al desarrollo de herramientas analíticas útiles.
Las técnicas de DL, por otro lado, requieren grandes volúmenes de datos (millones de puntos de datos, con miles de descriptores para cada punto) y requieren infraestructura de hardware especializada, como unidades de procesamiento gráfico (GPU), para realizar los cálculos necesarios. Un ejemplo típico de aplicación de DL es el análisis de imágenes, donde esta tecnología prospera.
En comparación con otros sectores (como el financiero), la IA ha llegado tarde al campo de la Biología, principalmente debido a la falta de infraestructura
tecnológica y personal especializado fuera de la industria. La formación de estos expertos requiere supervisión de personal competente, lo que se ha convertido en un cuello de botella en el desarrollo de la IA en este ámbito. Incluso si contáramos con la infraestructura y el conocimiento necesarios, la disponibilidad de datos seguiría siendo un factor limitante.
No todas las áreas de la Biología pueden beneficiarse igualmente de los avances en estas técnicas. La disparidad en la disponibilidad de datos es significativa. Por ejemplo, en UniprotKB hay disponibles unos 280 millones de secuencias de proteínas, pero sólo se han curado unas 848.000 interacciones en ~143.000 proteínas, según IntAct. Esto convierte a UniprotKB en un buen escenario para usar IA, gracias a las técnicas de secuenciación masiva, mientras que IntAct no es tan adecuado.
Es innegable que la IA ha demostrado un potencial transformador único, como lo demuestra la concesión del Premio Nobel de Química 2024 al diseño de proteínas artificiales funcionales y a los algoritmos de IA para la
predicción de estructuras terciarias de proteínas. Estos métodos avanzados han llegado para quedarse y evolucionan a un ritmo exponencial. Cada semana se publican nuevos modelos, incluidos modelos fundacionales y modificados. Entre ellos, los modelos de lenguaje para proteínas, derivados del procesamiento del lenguaje natural (NLP), han demostrado ser especialmente poderosos, como hemos presenciado en el último ejercicio de evaluación de métodos de predicción de función (CAFA5) presentados en ISMB2024. De entre los 10 mejores, 9 se basan en modelos de lenguaje.
Sin embargo, estos métodos presentan desafíos importantes. La escalabilidad de la producción de datos para entrenar modelos fundacionales sigue siendo un problema, y la interpretabilidad de estos modelos —auténticas “cajas negras”— ha dado origen a un campo emergente: la Inteligencia Artificial Explicativa (XAI, por sus siglas en inglés).
En conclusión, la IA está aquí para quedarse y su poder transformador será cada vez más evidente. No obstante, ¿Podremos aprovechar estos avances generados en la industria tecnológica de manera libre y transparente? ¿Queremos repetir situaciones como la de AlphaFold, cuyo código no ha sido liberado para el uso de la comunidad y el beneficio de la sociedad con uso restringido hasta meses después de su publicación?
En este número de la Revista SEBBM, hemos seleccionado cuatro artículos que creemos transmiten el mensaje que queremos compartir.
La Dra. Fátima Sánchez Cabo y sus colaboradoras Lucía Sánchez García e Inés Rivero, del CNIC, presentan aplicaciones de la IA en biomedicina, particularmente en medicina de precisión, e
introducen conceptos de inferencia causal y los avances de la IA generativa y explicativa.
El Dr. Pablo Chacón Montes (coordinador de la conexión CSIC de Biología computacional y Bioinformática, BCB.Hub) del IQF ‘Blas Cabrera’, CSIC, explica cómo la IA ha transformado el ámbito de la bioinformática estructural y describe el desarrollo de los modelos de lenguaje en la predicción de estructuras.
El Dr. Miguel A. Fortuna (BCB. Hub) y su colaborador Francisco J. Borrallo-Vázquez, ambos en EBD, CSIC, describen aplicaciones
de IA generativa en la genómica sintética, en particular para la creación de gemelos digitales usando Redes Generativas Antagónicas (GANs).
Los Drs. Ildefonso Cases (BCB.Hub), Ana Rojas (BCB.Hub), ambos en CABD (CSIC), la Dra. Rosa Fernández (BCB.Hub) y su colaboradora Gemma Martínez Redondo, ambas en IBE, CSIC, describen el problema de la anotación funcional de proteínas y la aplicación de modelos de lenguaje aplicados a proteínas para anotar funciones de genes desconocidos en millones de secuencias.

LA INTELIGENCIA
ARTIFICIAL COMO MOTOR
DE LA MEDICINA DE PRECISIÓN: PREDECIR Y EXPLICAR PARA SIMULAR
Lucía Sánchez García, Inés Rivero, Fátima Sánchez-Cabo
Centro Nacional de Investigaciones Cardiovasculares (CNIC), Madrid
https://doi.org/10.18567/sebbmrev_222.202412.dc1
La curiosidad científica anhela entender cómo funcionan las cosas. La pura predicción, si no somos capaces de entender los mecanismos subyacentes, nos suele dejar insatisfechos. En biomedicina, entender cómo funcionan los organismos es la base para poder proponer nuevas dianas terapéuticas y mejorar los tratamientos, la base para poder prevenir y curar. Los últimos avances en Inteligencia artificial (IA) demuestran cómo precisamente esta metodología puede ser clave para explicar los procesos que subyacen a la transición de la salud a la enfermedad a partir de las grandes cantidades de datos que se están generando, acelerando el conocimiento biomédico hasta cotas antes impensables.
La IA se utilizó inicialmente en biomedicina como elemento técnico para procesar desde imágenes hasta datos de electrocardiogramas. En este año, el premio Nobel a Hassabis y a Jumper (DeepMind) y a Baker ha demostrado que la aplicación de la IA a la biomedicina trasciende a la de ser una mera técnica. Además, el uso de técnicas de IA como agentes inteligentes que ayuden en la toma de decisiones en la práctica clínica es ya una realidad, aunque sigue siendo tomado con cautela, dada la suspicacia que generan las predicciones que no podemos explicar.
En este artículo mostraremos algunos de los últimos avances que demuestran cómo la IA, y en concreto la IA explicativa, se está convirtiendo en uno de los principales motores de la investigación biomédica, haciendo que la medicina de precisión pase de promesa a realidad.
Comenzamos un poco tarde…
Aunque la Inteligencia artificial lleva años utilizándose en banca, marketing o logística, su uso generalizado en biomedicina se retrasó hasta comienzos del siglo XXI. Las razones de este retraso han sido varias:
• En primer lugar, la disponibilidad de las grandes cantidades de datos cuantitativos y exhaustivos a nivel molecular, celular y de organismo en grandes poblaciones, necesarios para entrenar estos sistemas, era una quimera hasta hace poco más de una década. El abaratamiento de las técnicas ómicas, unido a la filosofía de compartición de datos potenciada por las agencias financiadoras, con el fin de maximizar los recursos, ha sido esencial en este cambio. Iniciativas pioneras como The Cancer Genome Atlas (TCGA), the Gene Expression Omnibus (GEO) o el
UK Biobank, por nombrar sólo algunas, han sentado las bases para poder utilizar millones de datos estandarizados en el entrenamiento de algoritmos de aprendizaje máquina en biomedicina.
• La dificultad de acceso a sistemas de computación de alto rendimiento en el entorno de la investigación pública, así como el coste inasumible del personal de administración de estos sistemas, normalmente muy escasos por su alta demanda en otros sectores que ofrecen más estabilidad y mejores condiciones económicas. En este sentido, la gran mayoría de los países ofrecen redes públicas de computación científica, como es el caso de la RES (Red Española de Supercomputación) en nuestro país. Sin embargo, el monopolio y la altísima demanda de los componentes necesarios en
supercomputación, como por ejemplo las graphical processing units (GPU), genera una gran incertidumbre acerca de cómo de realista será el uso de la supercomputación basada en recursos propios en la investigación biomédica pública, con las grandes corporaciones intentando copar este mercado.
• Finalmente, ha existido tradicionalmente una gran desconfianza hacia estas técnicas por parte de la comunidad científica biomédica, tachándolas de “caja-negra” o de “moda”. Esta desconfianza es algo normal, e incluso positiva para garantizar el rigor y la validez de los hallazgos que propicia. Descubrimientos que a menudo son difíciles de evaluar utilizando los controles tradicionales de revisión científica, debido al grado de abstracción que representan y la imposibilidad de revisar todos los desarrollos
en los que se basa de manera exhaustiva y su complejidad técnica. Y a la falta de personal formado en la materia trabajando en investigación biomédica, sobre todo en el sector público. Como en otros ámbitos, su aplicación generalizada tiene que ir de la mano de la definición de un marco ético y legal que está comenzando a clarificarse. Pese a todos estos problemas iniciales, el uso generalizado de la IA en Biomedicina es ya una realidad. Y el futuro es aún más prometedor.
Modelos predictivos para la medicina de precisión: De Mammaprint© a los scores poligénicos
Estratificar a los pacientes y a la población sana en función de su riesgo para mejorar su tratamiento, es uno de los objetivos de la medicina personalizada. La inferencia estadística clásica ha sido la herramienta utilizada para resolver

este problema durante casi un siglo, utilizando distintos tipos de regresión tanto univariante como multivariante. Estos métodos, sin embargo, tienen dificultades en la selección de predictores si existe un gran número de ellos a considerar, sobre todo en situaciones de interdependencia. La "maldición de la dimensionalidad" es un término a menudo utilizado para los modelos predictivos con datos ómicos, en los que el número de predictores (expresión génica o proteica, variantes genéticas, etc.) excede con mucho al número de individuos sobre los que realizar la inferencia. En este contexto, Laura Van´t Veer definió en 2003 en un artículo seminal en el campo la primera firma predictiva de recaída en 117 pacientes con cáncer de mama utilizando datos de expresión génica medida por microarrays. El tamaño muestral, que hoy parece ridículo, fue un hito en aquel momento. La firma estaba compuesta por 70 genes. Poco después, diversos bioinformáticos criticaron la metodología utilizada, dado que era posible encontrar muchos otros conjuntos de 70 genes diferentes con el mismo poder predictivo. Pese a ello, el estudio de Van´t Veer se validó en numerosos contextos clínicos y con un número de pacientes mucho mayor, llegando a convertirse en uno de los primeros casos de diagnóstico genético aprobados por la FDA. Hubo otros trabajos similares en aquella época, como la definición del Immunoscore© para la estratificación de los pacientes con cáncer de colón en base al tipo, la densidad y la localización de los linfocitos T infiltrados en los tumores.
El reducido número de participantes utilizado en estos y otros estudios similares a principios del siglo XX, cuando técnica y económicamente realizar test moleculares a gran escala resultaba muy costoso, demostraron el poder
predictivo de estas herramientas, aunque se necesitaron muchos estudios más para validarlos. Hoy en día, la disponibilidad de grandes cantidades de datos moleculares para grandes cohortes de individuos hace posible definir scores de riesgo de forma mucho más precisa, utilizando metodologías de aprendizaje máquina más robustas. Frente a la estadística clásica, los métodos de aprendizaje máquina como las redes elásticas, las redes neuronales, el gradient boosting o los random forest, ofrecen un marco conceptual más adecuado para la selección automática de los mejores predictores en situaciones de colinealidad. Pese a su potencia, es importante recalcar que estos métodos no pueden aplicarse a cualquier conjunto de datos, y que el “optimismo” en sus predicciones debido al sobreajuste, depende del número de predictores disponibles por cada caso a predecir. En un gran artículo de recomendada lectura, van der Ploeg y colegas utilizaron datos simulados para cuantificar el grado de optimismo en las predicciones de los diferentes algoritmos en base al número de casos por predictor. Actualmente, el uso de los algoritmos de aprendizaje automático utilizando los datos moleculares generados en grandes cohortes potenciados por los gobiernos de todo el mundo (UK Biobank, All of Us, IMPACT, entre otros) está permitiendo mejorar nuestra comprensión de enfermedades complejas, del cáncer a las enfermedades cardiovasculares y neurodegenerativas, siendo una pieza esencial en la medicina de precisión. Como ejemplo, los scores poligénicos (PGS) utilizan modelos de aprendizaje máquina para resumir el fondo genético heredado que contribuye a distintas condiciones fisiológicas y patológicas complejas. El catálogo de PGS recoge más de 5.000
PGS asociados con 655 rasgos. Utilizando metodologías similares, el grupo de Mike Inouye en la Universidad de Cambridge ha desarrollado modelos que predicen la expresión de cualquier gen o proteína en base a los SNPs de un individuo (https://www.omicspred.org/). Estos modelos han sido controvertidos, al dejar fuera la componente ambiental. Son, sin embargo, un esfuerzo valiente y en muchos casos útil, que abre la puerta a ser capaces de cuantificar el multi-oma de los individuos mediante modelos in silico, lo que permitiría abaratar los costes. En la misma línea se están desarrollando relojes epigenéticos mediante la aplicación de algoritmos de aprendizaje máquina que predicen la edad cronológica o la supervivencia (dependiendo de los modelos) a partir de los datos de metilación de una persona. Una vez más, al escepticismo general con los primeros relojes inferidos por Horvath le siguió una ola de curiosidad. Es cierto que aún no se ha demostrado por qué las CpGs que constituyen los relojes son las predictivas de la supervivencia y no otras, o por qué cada reloj contiene unas distintas. Más trabajo es necesario en esta línea para convencer realmente a la comunidad acerca de su validez.
Inferencia Causal e IA generativa: De la predicción a la explicación
La causalidad y la predicción se perciben a menudo como términos opuestos. Sin embargo, ambos se ocupan de la estimación de la misma probabilidad P(Y|X), con una diferencia que parece sutil pero que tiene grandes implicaciones a nivel técnico y de generalización: Los modelos predictivos predicen un resultado en base a observaciones, mientras que en inferencia causal el escenario en el que se realiza la predicción puede ser hipotético y no haber sido observado. Por esa

Figura 1
Esquema de la arquitectura de las redes neuronales generales y de los modelos generativos más utilizados actualmente
razón, la inferencia causal está ganando cada vez más atención y ayuda a la IA con la generalización de sus resultados. A su vez, algunos de los algoritmos computacionales desarrollados para resolver el problema de predicción pueden adaptarse para aproximar la probabilidad posterior de los datos a problemas complejos en los que los enfoques tradicionales no son capaces de encontrar una forma analíticamente cerrada.
Los modelos generativos profundos (Deep Generative Models, DGM) son marcos estadísticos que simulan nuevas observaciones de una variable de interés X, utilizando su función de verosimilitud Pθ(X), cuyo conjunto de parámetros θ se infiere de los datos observados. En problemas complejos, con gran cantidad de variables de confusión y de alta dimensionalidad, esto se vuelve cada vez más difícil, siendo la
inferencia un problema computacional "duro", denominado NP-hard Los modelos generativos, inspirados en la estadística bayesiana, obtienen muestras de una distribución latente Z Pθ(z) más sencilla y de menor dimensionalidad que X, y relacionan ambas variables mediante la distribución condicional Pθ(X|Z). Utilizando este marco, la probabilidad de los valores particulares de X se pueden estimar como Pθ(x) =∫ Pθ(x|z)Pθ(z)dz. Inferir esas distribuciones con técnicas de estadística tradicional como Expectation Maximitation (EM) es sumamente complicado e incluso imposible en el contexto biomédico, en el que hay un altísimo número de variables inter-relacionadas, algunas de ellas no conocidas y con una jerarquía predefinida, lo que da lugar a relaciones no lineales entre los predictores y las variables a predecir. Las redes neuronales
se han utilizado tradicionalmente como aproximadores naturales de funciones complejas, como las funciones de probabilidad que nos ocupan.
Los codificadores automáticos variacionales (VAE) y las redes generativas adversarias (GAN) se han vuelto cada vez más importantes en la inferencia variacional como habilitadores de la inferencia causal. Estos modelos están siendo muy utilizados para el análisis de datos a nivel de célula única, con aplicaciones desde el clustering automático de las células a la inferencia de redes de regulación. Un resumen de los principales algoritmos basados en DGMs para datos de scRNA-Seq se puede consultar en: https:// bioinfo.cnic.es/scDGMs/. La Figura 1 resume la arquitectura de las redes neuronales generales y de los modelos generativos más utilizados actualmente.
DOSIER CIENTÍFICO
Y de la predicción a la simulación
El uso de los DGM como forma de aproximar las complejas distribuciones posteriores que modelan las redes de causalidad subyacentes a la transición de la salud a la enfermedad, abre la puerta realmente a la comprensión global y no sesgada de los sistemas biológicos a través del análisis integrativo de datos multi-modales de alta dimensionalidad, no solo de datos moleculares (ómicos).
En un trabajo seminal, BIMMER trata de inferir las redes causales entre 405 fenotipos utilizando datos de los participantes del UK Biobank. Aunque la algorítmica utilizada se basa en Randomización Mendeliana, la aproximación a las funciones y la computación requeridas hacen necesaria la inferencia mediante técnicas de IA. Sólo así es resoluble un problema tan complejo como relevante.
Herramientas como BIMMER aplicadas sobre datos de grandes cohortes de población sana o de pacientes de distintas enfermedades, abren la puerta a la posibilidad de modelar los sistemas biológicos a gran escala, incluso a nivel de organismo completo. Con estas redes subyacentes modeladas gracias a la potencia de las nuevas metodologías de IA para resolver problemas de Inferencia Causal, se plantea un marco formal muy prometedor para simular el efecto de intervenciones en salud, mediante perturbaciones concretas del sistema por medio de gemelos digitales de cada individuo (Figura 2). Estos marcos in silico, si están suficientemente validados, permitirán a medio plazo optimizar la experimentación animal, así como reducir los tiempos en los ensayos clínicos.
Los retos que vienen
Con un panorama tan prometedor, parece que nada puede detener el imparable impacto de la IA en biomedicina. Sin embargo, las razones que impidieron un despliegue más rápido de estas metodologías a principios de siglo siguen estando ahí, algunas de ellas incluso más acentuadas. En primer lugar, la computación que se requiere para ejecutar estos algoritmos depende de tecnología copada por grandes empresas tecnológicas. La demanda es desproporcionada y corremos el riesgo de no poder hacernos con esta computación, no por falta de recursos económicos, sino por decisiones estratégicas de las empresas productoras. En este sentido, es esencial seguir apoyando las redes de colaboración computacionales para compartir los recursos. Por otro lado, los recursos humanos especializados son

Figura

cada vez más escasos. Los programas de financiación tanto nacional como internacional para captar talento están en marcha, pero sigue siendo complicado atraer al sector de la investigación pública a los especialistas. Es necesario que la sociedad perciba la ciencia como algo prioritario a lo que tenemos que contribuir entre todos. Además, tenemos que seguir avanzando en las políticas de compartición de datos que mantengan la integridad y privacidad de los mismos. Distintas infraestructuras europeas como ELIXIR, GDI, EHDEN o EUCAIM están contribuyendo a realizar de manera ordenada ese proceso esencial para el avance de la IA en biomedicina, que tantos frutos está dando. Finalmente, es esencial trabajar en formas de validar estos sistemas automáticos, una tarea que a menudo es mas compleja que la definición del propio modelo, pero que es esencial para mejorar la confianza en el uso de la IA en biomedicina y su posterior traslación a la práctica clínica.
Para saber más
Brown BC, Knowles DA. “Phenome-scale causal network discovery with bidirectional mediated Mendelian randomization”. bioRxiv 2020.06.18.160176. https://doi.org/10.1101/2020.06.18.160176
Ein-Dor L, et al. “Outcome signature genes in breast cancer: is there a unique set?”. Bioinformatics 21 (2005) 171–178. https://doi. org/10.1093/bioinformatics/bth469
Galon J, et al. “Type, Density, and Location of Immune Cells Within Human Colorectal Tumors Predict Clinical Outcome”. Science 313 (2006) 1960-1964. DOI: 10.1126/science.1129139
Rivero-García I, et al. “Deep generative models in single-cell omics”. Computers in Biology and Medicine 176 (2024) 108561. https://doi. org/10.1016/j.compbiomed.2024.108561
van der Ploeg T, et al. “Modern modelling techniques are data hungry: a simulation study for predicting dichotomous endpoints”. BMC Medical Research Methodology 14 (2014) 137. https://doi. org/10.1186/1471-2288-14-137
van ‘t Veer L, et al. “Gene expression profiling predicts clinical outcome of breast cancer”. Nature 415 (2002) 530–536. https://doi. org/10.1038/415530a
LA REVOLUCIÓN DE LA
INTELIGENCIA ARTIFICIAL EN LA BIOLOGÍA ESTRUCTURAL DE PROTEÍNAS
Pablo Chacón Montes
Instituto de Química Física Blas Cabrera (IQF ‘Blas Cabrera’), CSIC, Madrid
Dpto. Química Física Biológica Grupo de Bioinformática Estructural
https://doi.org/10.18567/sebbmrev_222.202412.dc2
La Inteligencia artificial (IA) ha revolucionado la biología estructural en tiempo récord. La capacidad de predecir con cierta precisión la disposición espacial de una proteína a partir de su secuencia nos permite explorar el espacio estructural de formas antes inimaginables, acelerar su caracterización experimental, y abrir nuevas ventanas para el desarrollo de herramientas computacionales capaces de diseñar proteínas que van más allá de lo explorado por la evolución natural. Las IA generativas, como los modelos de lenguaje y los procesos de difusión, han demostrado su capacidad para generar nuevas proteínas con propiedades específicas, alcanzando un éxito experimental notable. En este momento disruptivo y dinámico, en el que surgen a diario nuevas herramientas y aplicaciones, este artículo se centra en la estructura de proteínas, y excluye otros campos relacionados como el diseño de fármacos y ácidos nucleicos, donde la IA también es uno de los principales motores de avance.
AlphaFold es un ejemplo más de cómo la IA está transformado nuestro modo de vida, en este caso, revolucionando la biología estructural, un campo clave en el avance de la biología, la biotecnología y la investigación biomédica durante las últimas cinco décadas. En diciembre de 2020, AlphaFold 2 (AF2), desarrollado por Google-DeepMind, resolvió uno de los mayores desafíos de la biología: la predicción de la estructura tridimensional de las proteínas a partir de su secuencia de aminoácidos con una precisión comparable, en muchos casos, a la de las técnicas experimentales. Utilizando redes neuronales profundas, esta herramienta emplea la información de las estructuras conocidas y la co-evolución recogida en sus secuencias homólogas para predecir la estructura 3D de las proteínas. Seis meses después, la herramienta fue publicada en código abierto y permitió a la comunidad científica validar su eficacia (y limitaciones), provocando un impacto general y profundo (p. ej. la publicación original acumula más de 18.000 referencias) que ha culminado con la concesión en tiempo récord del Nobel en química para sus principales autores Demis Hassabis y John M. Jumper, junto a David Baker el gran pionero en modelado y diseño de proteínas que también tiene su propio desarrollo similar a AF2, RoseTTAFold. El uso de estas herramientas ha permitido expandir tres órdenes de magnitud la información estructural que disponemos. La base de datos AlphaFold DB (https://alphafold. ebi.ac.uk) ya almacena más de 214 millones de estructuras predichas por AF2, incluyendo el proteoma humano y de otras 47 especies, en contraposición con las 200.000 estructuras resueltas experimentalmente almacenadas en el Protein Data Bank (PDB). Al tiempo que intentamos

racionalizar esta ingente información estructural, su potencial es evidente como también que se nos presentan grandes retos que afrontar. Por ejemplo, para la identificación de proteínas estructuralmente similares en una base de datos de estas dimensiones se ha tenido que desarrollar una herramienta de búsqueda ultra-rápida (Foldseek) de nuevo con elementos de IA ante la imposibilidad de aplicar métodos tradicionales. Es importante resaltar que la capacidad de generar buenas predicciones permite acelerar la propia determinación experimental de estructuras. Podemos utilizar las predicciones para generar construcciones más estables eliminado zonas potencialmente desestructuradas, para resolver la estructura más rápidamente por reemplazo molecular en cristalografía de rayos-X, y, de forma similar, en crio-microscopía electrónica su
localización dentro de los mapas de densidad facilita su interpretación a resolución atómica. Como pescadilla que se muerde la cola, la capacidad de aprendizaje de las herramientas de IA y su precisión, aumentará al disponer de más estructuras experimentales. Sin duda, AF2 y RF se han convertido en herramientas muy populares al permitir el acceso a información estructural de las proteínas en minutos a partir de su secuencia. Si no lo ha hecho ya, el lector puede comprobar lo fácil que es obtener una predicción, bien buscándola directamente en AlphaFold DB o bien en la nube donde podrá encontrar implementaciones optimizadas de AF2 como Colabfold, o RoseTTAFold. Además de la predicción 3D de la estructura, por cada amino ácido se obtiene una estimación de la fiabilidad en su predicción, llamada pLDDT, que nos indica qué partes

de la estructura pueden ser fiables y cuáles no. Valores de pLDDT>90 corresponden a regiones predichas con precisión, valores entre 90 y 70 suelen corresponder a una predicción donde la cadena principal está bien modelada pero las cadenas laterales pueden contener algún error, y valores pLDDT<50 caracterizan zonas cuya predicción no es fiable, y suelen corresponder con regiones desordenadas. Cuando visualizamos una de estas predicciones, solemos ver que la mayoría son regiones con alta fiabilidad, más o menos salpicada con regiones de menor precisión muchas veces localizadas en la superficie en regiones flexibles como bucles. También son frecuentes zonas de baja precisión en el N y C terminales en forma de espagueti, que son particularmente extensas en organismos eucariotas más evolucionados. En general, los modelos de AlphaFold o RoseTTAFold son muy buenos, pero no siempre lo son, e incluso las regiones de alta fiabilidad pueden contener errores. En la red se pueden encontrar
un gran número de predicciones erróneas, que no invalidan la bondad y el carácter generalista de estas aproximaciones, pero que deben encender una luz amarilla de precaución sobre todo cuando queramos utilizar esas predicciones, por ejemplo en simulaciones atomísticas.
Aunque la entrada principal para la red neuronal de AlphaFold2 es una secuencia, en el primer paso se genera un alineamiento múltiple (MSA) buscando en distintas bases de datos secuencias similares a la de entrada. Precisamente, la calidad del alineamiento es uno de los factores determinantes para obtener una predicción más o menos precisa de la estructura. Un MSA diverso y profundo, con cientos o miles de secuencias alineadas, ayudará a identificar señales coevolutivas y utilizarlas para averiguar la correcta estructura 3D de la proteína. Por el contrario, un MSA poco profundo con pocas secuencias y baja variabilidad entre ellas, aumentará la posibilidad de errores en la predicción por la pobre señal coevolutiva.
Otras limitaciones incluyen la dificultad de modelar cambios conformacionales e interacciones proteína-proteína.
Potentes modelos de IA han cambiado el panorama de la comprensión del lenguaje, la maravillosa generación de lenguaje de ChatGPT no deja de asombrarnos. Precisamente, el éxito de los modelos de lenguaje de gran tamaño (LLM por sus siglas en inglés) en procesamiento del lenguaje natural (PLN) y la similitud entre nuestro lenguaje y el «lenguaje de las proteínas» motivaron el desarrollo de los modelos del lenguaje de las proteínas (PLM). Estos modelos, en lugar de aprender de las distribuciones de palabras/frase/textos, aprenden de las distribuciones de aminoácidos/secuencias/funciones. Los modelos PLM tratan las secuencias como datos de entrada de forma similar a como se trata el texto en modelos de PLN como GPT (Generative Pre-trained Transformer) o BERT (Bidirectional Encoder Representations from Transformers). El modelo de lenguaje ESM-2 desarrollado por
Meta y basado en BERT, entrenado con 250 millones de secuencias de proteínas, fue capaz de aglutinar y codificar a distintos niveles jerárquicos sus distintas propiedades (relaciones coevolutivas, propiedades bioquímicas y biofísicas de los aminoácidos, etc.) captando las complejas reglas que rigen la “gramática” de las proteínas. Tras el entrenamiento, este modelo de lenguaje puede transformar cualquier secuencia de proteínas en un vector que encapsula estas propiedades denominado embeddings
Los embeddings son una forma de representar la información de un texto en vectores para que los modelos de aprendizaje automático puedan medir la similitud de distintos textos y documentos en función de su significado, y así poder establecer relaciones que faciliten búsquedas, clasificaciones, etc. En este caso, los embeddings de ESM-2 son vectores que contienen información relevante de la secuencia como la conservación evolutiva, relaciones funcionales, motivos estructurales, etc. Utilizando modelos de aprendizaje profundo sobre estos embeddings de proteínas, se han logrado resultados espectaculares en la predicción de contactos de largo alcance, efectos mutacionales, de función e incluso también en la predicción de la estructura tridimensional. Esta última aplicación, denominada ESMFold, aunque no llega a los niveles de precisión de AF2, es muchísimo más rápida, lo que permite predecir eficazmente un ingente número de proteínas sin grandes recursos y puede ser una alternativa a AF2 en casos donde el MSA sea limitado. Hace pocos meses se ha presentado una nueva versión, ESM3, que es el primer modelo generativo que razona simultáneamente sobre la secuencia, la estructura y la función de las proteínas. Mejora a su antecesor en muchos aspectos:
su capacidad de predicción rivaliza con AF2, permite cierta especialización ya que está entrenado con diversos organismos y biomas, y permite diseñar proteínas con funcionalidades específicas. Como ejemplo ilustrativo de diseño con ESM3, han presentado una nueva proteína verde fluorescente (GFP) a partir de las estructuras y de los residuos críticos que definen su función. De los miles de diseños generados, se identificó uno con una similitud del 58% con la proteína fluorescente conocida más cercana con una fluorescencia similar a la observada en medusas y corales (Figura 1). Este resultado muestra la capacidad de ESM3 para explorar espacios proteicos que la naturaleza necesitaría millones de años de evolución en descubrirlos, y su potencial en la generación de nuevas proteínas funcionales.
En lugar de predecir cómo una secuencia se pliega, el aspecto crucial en el diseño de novo es resolver el problema inverso: diseñar una secuencia que, cuando se sintetice y pliegue, adopte la estructura específica con la función que deseamos. De los enfoques iniciales que estaban basados en modelos físicos (p. ej. Rossetta) hemos pasado al desarrollo de herramientas más potentes de aprendizaje profundo basadas en grafos (GNNs por sus siglas inglés). En este tipo de redes neuronales cada átomo o residuo del esqueleto proteico queda definido por un nodo que está conectado con los nodos cercanos a una distancia dada, que forman las aristas del grafo. En cada nodo de la red podemos añadir información como el tipo de aminoácido o las coordenadas tridimensionales, mientras que

Figura 1
DOSIER CIENTÍFICO
en las aristas se pueden incluir vectores que describen la distancia, dirección y orientación entre nodos conectados. Mediante el paso de mensajes, en el que cada nodo envía y recibe mensajes basados en las características de sus nodos y aristas vecinos, la red va agregando la información local y aprendiendo patrones complejos entre estructura y secuencia. ProteinMPNN es el paradigma de este tipo de redes, y su capacidad para predecir secuencias de aminoácidos compatibles con una determinada estructura ha sido validada experimentalmente en multitud de escenarios como el diseño de proteínas solubles, de nuevas proteínas, rediseño de estructuras y la generación de complejos simétricos. En su nueva versión, LigandMPNN, añade pequeños ligandos y ácidos nucleicos al proceso de diseño, y añade casos de éxito al diseño de proteínas que interaccionan con gran afinidad y especificidad con moléculas no proteicas. Es importante resaltar que las validaciones experimentales de este tipo de métodos tienen un alto grado de éxito, entre el 1020% de las secuencias predichas se obtiene la estructura deseada. Por esa razón no es de extrañar que el número y variedad de aplicaciones de diseño de proteínas crezca a pasos agigantados. Sin embargo, para su aplicación necesitamos una estructura de entrada. En un paso previo necesitamos definir un esqueleto o boceto estructural inicial y asegurarnos que sea “diseñable”, i.e. que al menos una secuencia se pliegue en esa estructura. Aunque siempre podemos partir de una estructura conocida intentando mejorar sus propiedades (p. ej. estabilidad o solubilidad), o reciclarla para otros propósitos, de nuevo, la IA nos ofrece una solución con los modelos de difusión. Durante el entrenamiento
de estos modelos generativos de aprendizaje profundo, primero se añade ruido gaussiano a las estructuras de proteínas conocidas para luego entrenar la red para recuperar las estructuras originales mediante un proceso iterativo de eliminación de ruido. Así, el modelo aprende las distribuciones de probabilidad del espacio de estructuras proteicas, lo que le permite, en el momento de la generación, recibir ruido gaussiano y transformarlo de forma iterativa en nuevos esqueletos proteicos cuya secuencia tendremos que diseñar.
RFDiffusion, aprovechando los pesos preentrenados de RoseTTAFold y las capacidades de ProteinMPNN para resolver el problema inverso, obtiene resultados realmente espectaculares. Con esta herramienta se han generado nuevas arquitecturas de barriles alfa-beta visibles mediante CryoEM que superan las variaciones estructurales del clásico pliegue de barril TIM –un diseño de proteínas para cinco dianas proteicas terapéuticas, con una tasa de éxito del 18% con 95 diseños con especificidad picomolar-. La nueva versión RFdiffusionAA, que utiliza RoseTTAFold All-Atom, la versión mejorada de RF, expande el rango de aplicabilidad al diseño de proteínas que unen pequeños ligandos. Por ejemplo, ha diseñado proteínas que se unen a bilina, hemo y digoxigenina con estructuras muy diferentes a las proteínas que unen estos compuestos que se encuentran en el PDB. Además, los modelos de difusión pueden incorporar restricciones en generación que nos permiten incorporar propiedades y funciones deseadas en el diseño.
Chroma, otro modelo de difusión, también puede condicionarse con texto lo que abre la posibilidad de que en algún futuro tengamos una herramienta a la
que podamos decirle «Diseña una proteína pequeña, soluble y que se una a la proteína X». A pesar de estos increíbles avances queda mucho camino por recorrer, en particular, poder incorporar en el diseño la exploración del espacio conformacional y de interacciones, lo que nos permitiría crear proteínas que respondan a determinados estímulos o incluso generar nuevos mecanismos enzimáticos.
El impacto y el rango de aplicación de las herramientas basadas en IA (Figura 2) es impresionante. Muy recientemente, Google DeepMind ha publicado AlphaFold3 (AF3), que extiende su capacidad más allá de proteínas individuales con la predicción de sus interacciones con ácidos nucleicos, iones y pequeñas moléculas. A pesar de que reportan resultados muy prometedores que mejoran la capacidad y el rango predictivo de la versión anterior, de momento, AF3 sólo está accesible de forma limitada en un servidor web junto con unas draconianas licencias de uso. De igual forma ESM3, aunque el código sea accesible, prácticamente solo sus desarrolladores disponen de los recursos computacionales necesarios para su entrenamiento, e incluye una oscura licencia no comercial. Aunque estamos acostumbrados a que estas compañías controlen nuestra vida digital sabiendo donde estamos, lo que buscamos, compramos, etc., parece excesivo que vayan a controlar nuestra capacidad de investigación. Esperemos que la comunidad científica logre que recapaciten y vuelvan a la senda del código abierto, donde el Prof. D. Baker ha demostrado su activismo a lo largo de los años al permitir el acceso a sus herramientas de predicción y el diseño de proteínas, que desde aquí agradecemos.

2
Para leer más
Abramson J, et al. “Accurate structure prediction of biomolecular interactions with AlphaFold3”. Nature 630 (2024) 493–500. https://doi.org/10.1038/s41586-024-07487-w
Krishna R, et al. “Generalized biomolecular modeling and design with RoseTTAFold AllAtom”. Science 384 (2024) 2528. DOI: 10.1126/science.adl2528
Varadi M, et al. “AlphaFold Protein Structure Database in 2024: Providing structure coverage for over 214 million protein sequences”. Nucleic Acids Research 52 (2024) D368–D375. https://doi.org/10.1093/nar/ gkad1011
Watson JL, et al. “De novo design of protein structure and function with RFdiffusion”. Nature 620 (2023) 1089–1100. https://doi.org/10.1038/s41586-023-06415-8
Winnifrith A, et al. “Generative artificial intelligence for de novo protein design”. Current Opinion in Structural Biology 86 (2024) 102794. https://doi.org/10.1016/j.sbi.2024.102794
Figura
INTELIGENCIA ARTIFICIAL GENERATIVA (GENAI) PARA DISEÑAR GENOMAS DE ORGANISMOS DIGITALES
Francisco J. Borrallo-Vázquez y Miguel A. Fortuna
Laboratorio de Biología Computacional
Estación Biológica de Doñana (EBD), CSIC, Sevilla
https://doi.org/10.18567/sebbmrev_222.202412.dc3
La Inteligencia Artificial
Generativa (GenAI por sus siglas en inglés) es una rama de la Inteligencia artificial (IA) que se enfoca en la creación de nuevos contenidos, como imágenes, texto, música y otros datos, a partir de patrones y datos preexistentes. En lugar de simplemente analizar o clasificar información, los modelos de GenAI, como ChatGPT, aprenden a replicar y crear datos a partir de los que han usado durante su entrenamiento. Este tipo de algoritmos es particularmente útil en áreas creativas y de diseño, como la creación de arte digital, la generación de contenido en videojuegos, la composición musical y la simulación de conversaciones humanas. Pero además, la GenAI también tiene aplicaciones en ámbitos
científicos, como el diseño de moléculas activas novedosas en farmacología, la predicción de patrones de coexistencia entre especies en comunidades ecológicas, y la generación de secuencias genómicas en la investigación biomédica.
La genómica sintética busca diseñar y construir genomas completos para encontrar los principios fundamentales responsables de la función del genoma. Los avances recientes en ensamblaje, edición, y síntesis de ADN junto con las innovaciones computacionales en la GenAI han impulsado este campo. El objetivo de la investigación que llevamos a cabo con genomas digitales es, precisamente, guiar el diseño y construcción de genomas sintéticos de organismos naturales. Sin
embargo, trasladar lo aprendido in silico a los proyectos de genómica sintética sigue siendo un proceso altamente complejo. Nuestros esfuerzos en la aplicación de la GenAI a genomas de organismos digitales se centran en optimizar este proceso para intentar, en una segunda fase, trasladar lo aprendido al diseño, construcción, entrega, ajuste y aplicación de genomas sintéticos de organismos naturales.
Evolución digital
La evolución digital es una forma de computación evolutiva en la que programas informáticos autorreplicantes —organismos digitales— mutan y evolucionan dentro de un entorno computacional definido por el usuario. Avida es la plataforma de software más
utilizada para la investigación en evolución digital y se ha consolidado como el nexo de unión entre la simplicidad y abstracción de los modelos matemáticos, por un lado, y la complejidad y realismo de los experimentos en el laboratorio y en condiciones naturales, por el otro. Avida cumple con los tres requisitos esenciales para que tenga lugar el proceso evolutivo: replicación, variación heredable y eficacia biológica diferencial —fitnes-. Este último requisito emerge de la competencia por los recursos limitados de espacio en memoria (RAM) —donde “viven” los organismos digitales— y tiempo de la unidad central de procesamiento (CPU) que usan los organismos digitales para replicarse. Un organismo digital en Avida consiste en una secuencia de instrucciones —su genoma— y una CPU virtual que ejecuta esas instrucciones.
El espacio de secuencias que pueden codificar organismos digitales con genomas de longitud —número de instrucciones— L extraído de un alfabeto de instrucciones disponible A, comprende AL genomas diferentes. Si consideramos genomas con L = 100 instrucciones tomadas de un alfabeto de A = 26 instrucciones (el lenguaje genético de Avida; Figura 1), el espacio de secuencias es enorme: 26100 = 3,14 × 10141. Cualquier genoma en este espacio codificará un organismo viable si éste es capaz de autorreplicarse. Encontrar genomas viables en este gigantesco espacio mediante la generación de secuencias aleatorias es como buscar una aguja en un pajar. El coste computacional es muy alto. Por ejemplo, se necesitan más de un millón de secuencias aleatorias para encontrar un solo genoma viable de 100 instrucciones. Por tanto, necesitamos
métodos mucho más eficientes en la búsqueda de genomas que codifiquen organismos viables que puedan usarse como ancestros— wild type— a partir de los cuales iniciamos nuestros experimentos evolutivos destinados a desvelar los mecanismos responsables de la biodiversidad del planeta. Y es en este punto donde la GenAI entra en juego.
Redes generativas antagónicas
Las Redes Generativas Antagónicas (GANs por sus siglas en inglés) son una clase de algoritmos de GenAI que imitan la “carreras de armamentos” evolutivas que tienen lugar, por ejemplo, entre los depredadores y sus presas (donde el depredador evoluciona para ser más eficiente en la búsqueda y captura de su presa, y su presa evoluciona para evadir más fácilmente a su depredador). Una GAN consiste en dos

El lenguaje genético del genoma de los organismos digitales está formado por 26 códigos de instrucciones. Es decir, a diferencia de los cuatro nucleótidos que constituyen el ADN del genoma de los organismos naturales o de los 20 aminoácidos a partir de los cuales se forman las proteínas, cada posición del genoma de un organismo digital puede estar ocupada por una de las 26 “letras” posibles.
Figura 1
DOSIER CIENTÍFICO

Figura 2 avidaDB es una base de datos que contiene más de un millón de genomas de organismos digitales disponibles para ser usados en experimentos evolutivos. Con el fin de facilitar el acceso a avidaDB hemos desarrollado una librería para el lenguaje de programación R (avidaR) que permite hacer descargas y búsquedas selectivas de genomas.
redes neuronales artificiales con múltiples capas que se entrenan de manera concurrente: una red generativa y una red discriminativa. Dentro de nuestro propósito de encontrar genomas que codifiquen organismos digitales viables, la red generativa (generador) usa ruido aleatorio gaussiano para producir genomas tan reales (es decir, genomas que codifican organismos viables) como sea posible, de modo que se entrena para generar genomas falsos (es decir, que no codifican organismos viables). La red discriminativa (discriminador) recibe genomas aleatorios del generador y genomas reales de la base de datos avidaDB (Figura 2), y decide si el genoma es real o no. Tanto la red generativa como la discriminativa se entrenan a la vez, jugando una contra la otra para maximizar sus objetivos: el del generador es proporcionar genomas aleatorios lo suficientemente reales como para engañar al discriminador (que los clasificaría como reales aunque no lo sean), y el del discriminador es obligar al generador a producir genomas ni tan reales ni tan aleatorios como
para clasificarlos acertadamente como reales o falsos, respectivamente. Cuando este aprendizaje recíproco se equilibra, el generador proporciona genomas aleatorios con alta probabilidad de que codifiquen organismos digitales viables (Figura 3)
Una de las ventajas de usar esta aproximación computacional como gemelo digital del diseño de genomas de organismos naturales es que podemos validar el éxito del entrenamiento de las GANs inmediatamente y sin coste alguno. Es decir, a diferencia del diseño de secuencias genómicas mejoradas para la industria y la biomedicina que requieren posteriormente su síntesis química para corroborar que se ha implementado con éxito la funcionalidad biológica deseada, la comprobación de la viabilidad de los genomas de organismos digitales generados con las GANs se comprueba directamente en Avida. Sólo necesitamos ejecutar el código del organismo digital que contiene el genoma que proporciona el generador de la GAN y, en cuestión de milisegundos, observar si es o no capaz de replicarse.
Y esta validación retroalimenta a su vez el sistema de entrenamiento de la GAN porque los genomas generados que codifiquen organismos viables formarán parte de la base de datos de genomas reales —avidaDB— con la que se entrenan tanto el generador como el discriminador.
No sólo evolución sino también coevolución digital
En Avida, los organismos digitales necesitan consumir recursos del entorno computacional para replicarse, de forma análoga al consumo de nutrientes por parte de las bacterias. Los recursos computacionales definidos en Avida se consumen sólo si el organismo es capaz de realizar funciones matemáticas sencillas con números binarios mientras ejecuta las instrucciones que constituyen su genoma. Y de manera análoga a como la bacteria E. coli metaboliza glucosa o citrato como fuente de carbono, un organismo digital puede consumir el recurso asociado a realizar una suma (operación booleana OR) o una multiplicación (operación booleana AND). Esta

Figura 3
Una Red Generativa Antagónica (GAN) para el diseño de genomas consta de dos redes neuronales que se entrenan simultáneamente: una red generativa (generador), que usa ruido aleatorio para producir genomas aleatorios y se entrena para generar genomas falsos pero no tan aleatorios, y una red discriminativa (discriminador), que recibe genomas del generador y de la base de datos avidaDB, clasificándolos como reales o falsos. Ambas redes se entrenan una contra la otra para mejorar su precisión. Al equilibrarse este proceso, el generador produce genomas con alta probabilidad de codificar organismos digitales viables que, de serlo, pasarán a formar parte de la base de datos.
capacidad que tiene un organismo digital de realizar operaciones booleanas para consumir recursos define su fenotipo o funcionalidad. Esta propiedad funcional de los organismos digitales ha permitido implementar interacciones hospedador-parásito, en donde algunos organismos —parásitos— son capaces de “robar” la energía que necesitan (ciclos de CPU) para ejecutar las instrucciones de sus genomas a otros organismos digitales —sus hospedadores. Un parásito podrá infectar a un hospedador si realiza el menos una de las funciones booleanas que lleva a cabo éste. Es el equivalente digital al acoplamiento entre las fibras de la cola de un virus bacteriófago y los receptores de membrana de la bacteria. La presión selectiva que imponen los parásitos beneficiará a aquellos hospedadores que acumulen mutaciones en el
genoma que cambien la función booleana que realizan —el receptor de membrana bacteriano. De esta forma el hospedador podría escapar de los parásitos. Por tanto, si pensamos en el creciente interés que ha adquirido el uso de virus bacteriófagos como agentes terapéuticos en la lucha contra las infecciones bacterianas resistentes a los antibióticos, se requiere un método de generación de secuencias genómicas que posean la funcionalidad necesaria para infectar variantes genómicas que puedan emerger durante la evolución bacteriana (Figura 4)
De los genomas de organismos digitales a los genomas de organismos naturales El objetivo fundamental del uso de gemelos digitales, como Avida, es complementar los estudios llevados a cabo en el laboratorio. El
paso crucial es comprobar si lo que aprendemos estudiando organismos digitales en un ordenador es útil para entender los mecanismos y procesos que suceden en los organismos naturales. Y en este punto se puede ver el vaso medio vacío o medio lleno, es decir, fijarse únicamente en las diferencias entre organismos digitales y naturales (que las hay) o profundizar en las características comunes (que también las hay, y muchas). Para nosotros, el proceso evolutivo es independiente del substrato material que contiene y transmite la información, sea la química molecular basada en el carbono o el estado de los electrones en un semiconductor. Lo verdaderamente importante es que haya entidades replicantes (células o programas informáticos) que contengan información heredable (genoma) y que las variaciones en la información

Figura 4
Un parásito digital podrá infectar a un hospedador digital si realiza el menos una de las funciones booleanas que lleva a cabo el hospedador. Es el equivalente digital al acoplamiento entre las fibras de la cola de un virus bacteriófago (fago) y los receptores de membrana de sus hospedadores bacterianos. Una vez se produce ese acoplamiento, el parásito infectará a su hospedador. Este proceso impone una presión selectiva que beneficiará a aquellos hospedadores que adquieran mutaciones que les permitan realizar nuevas funciones booleanas, de manera análoga a cambios en los receptores de membrana de las bacterias. De esta forma, el hospedador podrá escapar de los parásitos. Por tanto, se require un método de generación de genomas de parásitos digitales que posean la funcionalidad necesaria para infectar a la variante genética específica que pueda emerger durante la evolución del hospedador.
que contienen les otorguen mayor o menor eficacia en la transmisión de esa información durante su proceso de replicación.
¿Hasta qué punto este enfoque digital puede ayudar a diseñar nuevos genomas de organismos naturales? Si nos centramos en el uso de fagos como agentes terapéuticos, podemos entrenar las GANs con los genomas de los fagos y los de sus hospedadores bacterianos para ayudar a los investigadores a identificar nuevos hospedadores susceptibles de ser infectados. De hecho, el grupo de Jim Collins en el MIT ha desarrollado recientemente una herramienta llamada BioAutoMATED, que utiliza modelos de lenguaje entrenados en secuencias genómicas biológicas para interpretar y diseñar genomas que posean una funcionalidad específica, análoga a las funciones booleanas que son capaces de llevar a cabo los parásitos digitales en Avida.
Para leer más
Eugene L, et al. “Relevant applications of generative adversarial networks in drug design and discovery: molecular de novo design, dimensionality reduction, and de novo peptide and protein design”. Molecules 25 (2020) 3250. https://doi.org/10.3390/ molecules25143250
Fortuna MA, et al. “The genotype-phenotype map of an evolving digital organism”. PLoS Computational Biology 13 (2017) e1005414. https://doi.org/10.1371/journal.pcbi.1005414
James JS, et al. “The design and engineering of synthetic genomes”. Nature Review Genetics (2024) https://doi.org/10.1038/ s41576-024-00786-y
Valeri JA, et al. “BioAutoMATED: an end-to-end automated machine learning tool for explanation and design of biological sequences”. Cell Systems 14 (2023) 525-542. https://doi.org/10.1016/j. cels.2023.05.007
Yelmen B, et al. “Creating artificial human genomes using generative neural networks”. PLoS Genetics 17 (2021) e1009303. https:// doi.org/10.1371/journal.pgen.1009303
BIOANALIZADORES


Máxima resolución y sensibilidad

Última tecnología en electroforesis capilar con detección por fluorescencia al alcance de todos

Genotipado
Producto de PCR (DNA/RNA)
Control de calidad NGS





CRISPR


Purificación de plásmidos





RFLP

Análisis SSR











Oligonucleótidos cfDNA gDNA







ANOTACIÓN DE FUNCIÓN EN PROTEÍNAS USANDO MODELOS DE LENGUAJE
Ana M Rojas Mendoza*e Ildefonso Cases
Centro Andaluz de Biología del Desarrollo (CABD), CSIC, Sevilla. Grupo de Biología Computacional y Bioinformática
Gemma Martinez-Redondo y Rosa Fernández
Instituto de Biología Evolutiva (IBE), CSIC, Barcelona. Metazoa Phylogenomics lab
https://doi.org/10.18567/sebbmrev_222.202412.dc4
El poder transformador de la Inteligencia artificial (IA) ha irrumpido en la biología estructural, mediante desarrollos relacionados con la predicción de la estructura terciaria (ahora de complejos de proteínas y ligandos tras meses de espera), y con el diseño de proteínas a la carta. Dado que la IA ha llegado tarde a la biología, uno esperaría que el impacto sobre todas las disciplinas del dominio biológico sea gradual. Lo que es evidente es que la IA ha llegado para quedarse, y es cuestión de tiempo que llegue a todas las áreas en las que pueda florecer.
La predicción de función es un problema bien conocido, persistente, y que sigue sin resolverse, pese a décadas de desarrollo y esfuerzos los métodos no progresan, representando un reto enorme de la Biología computacional, así que demos un repaso histórico y conceptual a la problemática.
¿Qué es función? Es un concepto subjetivo, muy dependiente de contexto, que normalmente extraemos de la literatura científica. Para organizar la información, hemos generado ontologías de genes (Gene Ontology), que incluyen términos GO (por sus siglas en inglés), que no son más que vocabularios controlados relacionando términos asociados a funciones. Estos términos se representan en una estructura de grafo donde los nodos del mismo son los términos, conectados por flechas que indican las relaciones entre los mismos. La raíz del grafo representa los términos más generales (por ejemplo, Proceso Biológico, etc.) y va progresando con términos más específicos si avanzamos de nodo en nodo. La profundidad en el grafo es variable denotando la especificidad del término, y no todas las ramas del grafo tienen la misma profundidad, ni todos los organismos tienen los mismos
grafos. Por ejemplo, los términos relacionados con desarrollo de ala en mosca no se encuentran en el grafo de la levadura.
Lo que anotamos en realidad es el producto codificante de los genes, las proteínas, y esas anotaciones se asocian de nuevo a los genes correspondientes. Por otro lado, “función” también es un concepto bioquímico, y en estructura de proteínas se asocia a sitios de unión o sitios catalíticos. Hay que decir que existe un sesgo en cuanto a los genes que se estudian, puesto que Stoeger et al., en 2018, publicaron un trabajo en el cual demuestran que estudiamos los mismos genes de manera circular, y que incluso en humanos, el foco cae en unos 2.000 genes de los 19.000 posibles. Atribuyeron causas multifactoriales, pero el mensaje es que no estamos progresando. En otras palabras: la información funcional de la que disponemos
es muy parcial y está muy sesgada por intereses específicos.
¿Cómo se asigna la función a una secuencia nueva? Pues usamos la similitud de secuencia (concepto matemático, cuantitativo) para identificar homología de secuencia (concepto cualitativo). Esta aproximación está basada en dos premisas, la primera se fundamenta en la evolución molecular y las relaciones evolutivas entre las secuencias de las especies, en particular los ortólogos y los parálogos (ver Ohno, 1970 y Zuckerkandl y Pauling, 1965). Recordamos que un ortólogo es un gen que estaba presente en el ancestro común de dos especies, y que cuando ocurrió el evento de especiación cada especie nueva adquirió una copia del gen, mientras que un parálogo es un gen que se duplica a partir del ortólogo tras el proceso de especiación (Figura 1A)
Y ¿cómo relacionamos esto con la función? Pues a través de la conjetura del ortólogo propuesta por Nehrt et al., en 2011, que sugiere que los ortólogos retienen la función, mientras que los parálogos (duplicaciones de un gen tras especiación) la diversifican, aunque estas nociones las introdujo E. Koonin en los 90. Es decir, asumimos que las secuencias ortólogas “realizan” la misma función. Sin embargo, uno de los errores conceptuales más profundos y persistentes en disciplinas como la biología del desarrollo o la genómica, es utilizar la similitud funcional entre dos genes para definir ortología, como ya discutió G. Theißen en 2002. La analogía funcional no implica ortología, ni la ortología implica necesariamente la analogía funcional.
Desde un punto de vista termodinámico, los estudios seminales de C. Anfinsen en los 60, llevaron a concluir que la estructura más
Figura 1

Premisas en las que basamos la asignación de función. A) Relaciones evolutivas a nivel molecular respecto a eventos de especiación. El ancestro común posee un gen (círculo negro) que se transfiere a cada especie tras un evento de especiación. En un caso, se duplica y en otro no. B) El paradigma de secuencia/estructura/función entre la estructura tridimensional y la secuencia, a mayor similitud de secuencia, mayor la de su estructura (el eje Y es rmsd, la distancia entre carbonos alfa de la estructura, mientras que el eje X es el % de identidad de la secuencia de aminoácidos). Siguiendo con la hipótesis de Anfinsen, el estado de energía más bajo es el más estable y el que se asocia a la función de la proteína. Hoy sabemos que esto no es así.
DOSIER CIENTÍFICO
Figura 2

Asignación de función. A) Transferencia de “función” basada en la similitud de la secuencia de aminoácidos. B) Problemas de asignación automática de función por trasferencia errónea (función azul). C) Problemas de asignación masiva a miembros de subfamilias de proteínas. D) Caso de metamorfismo en proteínas, que implica cambio radical de estructura cuando la proteína adopta diferentes funciones.
estable asociada a una secuencia se asociaba a una función fisiológica determinada (Figura 1B). Estas dos premisas han originado el siguiente paradigma: “una secuencia, una función, una estructura”, que es central a todos los métodos computacionales de predicción de función y predicción de estructura. Entonces, si dos secuencias de proteínas muestran una identidad mayor a un umbral (normalmente un 40%), que puede obtenerse usando métodos que estiman cómo de idénticas son dos secuencias en su composición (p. ej. BLAST), se transfiere toda la anotación simultáneamente de una a otra (Figura 2A). Esta transferencia es ruidosa, puesto que a veces se transfieren funciones que no están en la secuencia a identificar porque la región responsable de la función (imaginemos un dominio kinasa) no está presente en la secuencia que
queremos anotar (ver en Figura 2B, región azul). También puede ocurrir que se transfiera la misma función a miembros de familias de proteínas, que son aquellas que se dividen en subfamilias a distintos niveles de identidad de secuencia, pero por encima del umbral, que realizan funciones diferentes (Figura 2C). Además, hay numerosas excepciones que invalidan estas premisas, por ejemplo, hay ortólogos que diversifican función, mientras que hay parálogos que adoptan la función original del ortólogo. En otros casos, las proteínas tienen más de una función o la cambian como consecuencia de señales biológicas. Un caso muy llamativo de esto son las histonas, proteínas clásicas en el mantenimiento estructural de la cromatina, que se convierten en enzimas (reducen cobre) cuando se asocian en tetrámeros en ciertas condiciones. Y
no menos relevante, hay proteínas que cambian su estructura sustancialmente para cambiar de función, las llamadas proteínas “metamórficas” (Figura 2D) Por último, incluso asumiendo que el paradigma fuese cierto, hay miles de proteínas que no presentan homología de secuencia con ninguna otra en las bases de datos, especialmente aquellas que provienen de organismos no modelo, o que tienen una composición rara (baja complejidad), lo que hace que frecuentemente queden sin anotar.
Por lo tanto, el problema subyace en las premisas, no en los métodos, que son muy potentes, ni en los datos, cuya disponibilidad es enorme. Sin embargo, la mejora en la predicción se ha estancado, según los resultados publicados del último CAFA (Critical Assessment of Functional Annotation), la comunidad que
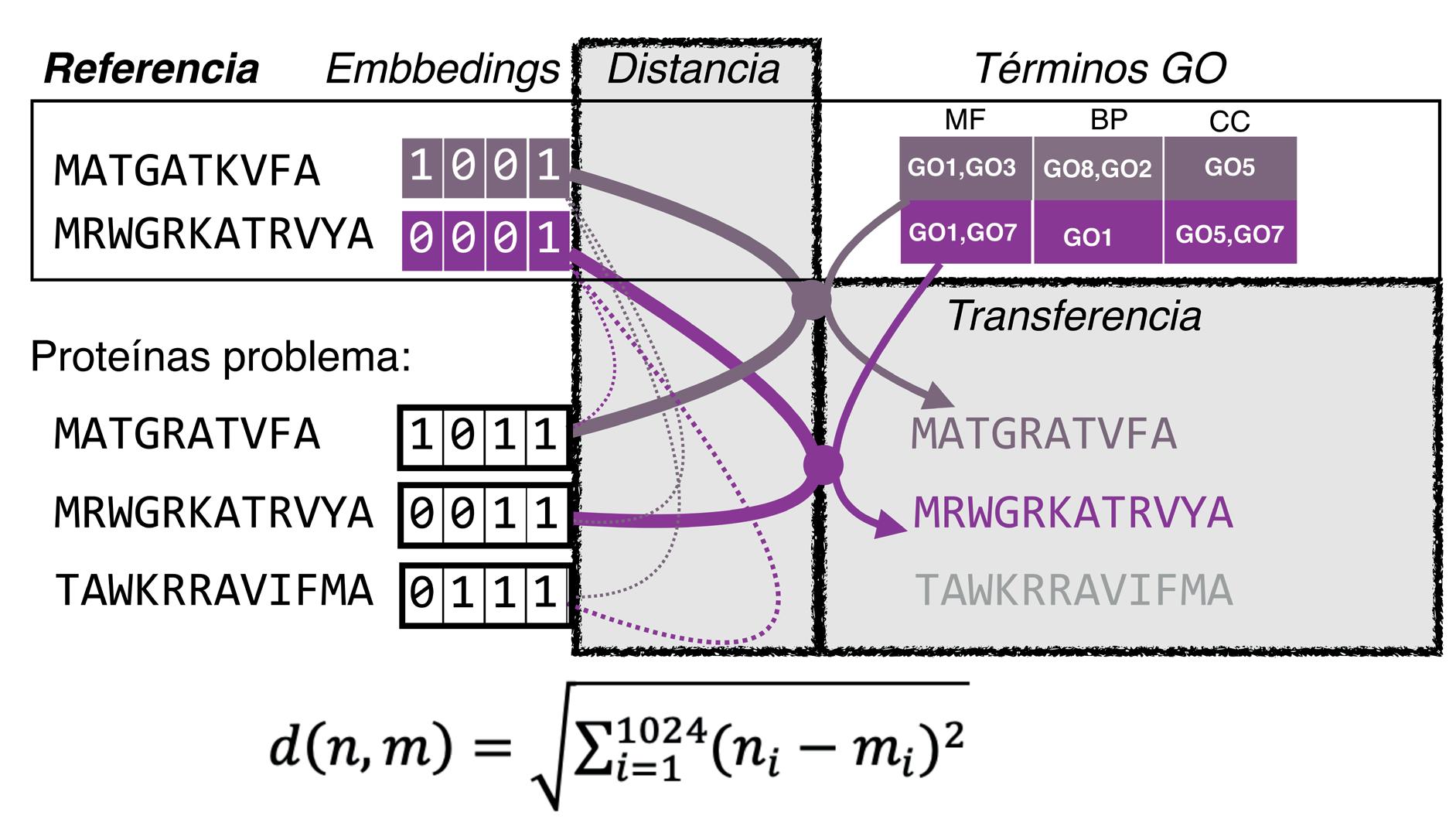
Figura 3
Asignación de función usando modelos de lenguaje. Las secuencias tanto de referencia como problema se codifican en embeddings mediante transformers, se calcula la distancia en el espacio de embeddings, y a distancias más pequeñas (líneas más anchas) se transfieren los términos GO de las secuencias de referencia.
evalúa los métodos de predicción de función*.
Entonces, ¿Cómo podemos proceder ignorando el paradigma?, usando aproximaciones que no requieran la homología de secuencia.
En este contexto, los métodos de IA basados en “transformers“ emergen como alternativas prometedoras, puesto que no usan información evolutiva.
Los modelos de lenguaje de proteínas (pLM) se han derivado de los modelos de lenguaje natural. En IA, igual que en el lenguaje natural, las frases tienen estructuras, palabras y reglas, y podemos considerar una secuencia de proteínas como una frase y extraer propiedades de esa frase. Hay muchos pLM (introducidos brillantemente en el artículo del Dr. Chacón en este mismo dosier), pero en este artículo nos centraremos en el modelo ProtTrans
publicado por Elnaggar et al., en 2022, que es el que más se ha utilizado para la anotación de función.
Estos modelos que se han adoptado del lenguaje natural, codifican las secuencias de amino ácidos en objetos matemáticos denominados embeddings, que son básicamente vectores. Littman et al., en 2021, fueron pioneros en implementar este método para transferir anotaciones, reemplazando la homología de secuencia por la distancia en el espacio de vectores (Figura 3), y usando el set de datos de función de CAFA3, que es un conjunto de datos curados de función, para validar su método. Sin embargo, siendo útiles, estos conjuntos tienen un carácter universal, que no permite el abordaje a nivel de organismo. Nosotros decidimos abordar estas limitaciones, y para ello en
un esfuerzo colaborativo nos planteamos resolver tres cuestiones fundamentales: ¿Cómo funcionan estos métodos a nivel de organismo? ¿Cómo de bien recuperan la función obtenida en experimentos de expresión génica? Y si los usamos a gran escala en organismos no modelo poco convencionales, ¿Podemos anotarlos con confianza y obtener información funcional relevante?
Para responder a la primera pregunta usamos organismos modelo, que sirven de buena referencia porque tienen anotaciones manuales muy trabajadas y revisadas por expertos (por ejemplo, ratón, mosca, gusano y levadura). Lo primero que nos sorprendió fue comprobar que el estado de anotación de estos organismos, no es completo. Por ejemplo, un 30% de los genes del gusano C. elegans carece de anotaciones, al igual que la mosca D. melanogaster, con un
17% aproximado de sus genes sin información funcional.
La mayoría de estos genes, o bien no presentan homología identificable con otras secuencias en las bases de datos, o bien estas secuencias presentan características particulares como complejidad, desorden, etc. La idea era reemplazar las anotaciones de referencia, que están bien definidas por las anotaciones predichas por los pLM, y compararlas.
Pero, ¿Cómo evaluamos si las predicciones son buenas? Usamos varias aproximaciones. La primera es valorar su “precisión”, es decir, cómo es el acierto de términos. Comparando cuántas veces un método de predicción acierta el término anotado, nos da una idea de la precisión. Otro aspecto a considerar es el “alcance” del método. Imaginemos que nuestro método fuese un arma automática, lo relevante es saber cuántas veces tendría que “disparar” para acertar los términos (los GO anotados de referencia). Si dispara mucho, por puro azar acertará alguna vez, pero fallará muchas veces si no es un buen método. Si dispara muy poco y acierta todas las veces, será un método muy preciso, pero con alcance limitado pues no acierta todo lo que podría acertar. Un método perfecto sería aquel que disparando todas las veces que debería disparar (el número real de anotaciones), acertara todas las veces en la diana.
Especialmente relevante, “cómo de informativo” es el acierto, en función de dónde esté el término en el grafo de GO. Si el método sólo acierta cerca de la raíz que es el término más general (p. ej. “Molecular Function”), entonces la información que proporciona no es detallada y no ayuda a discriminar funciones. Y, por último, evaluando “cómo de similar” es la información que produce podemos establecer si
el método está acertando como debería. Para ello, calculamos la similitud semántica de los términos predichos respecto a los anotados por expertos, que es una manera de medir si los grupos de términos asociados a cada proteína son similares, y en su caso, la similitud ha de ser alta para que el método sea fiable.
Estimando todos estos aspectos, concluimos que el modelo ProtTrans anota prácticamente todos los genes y produce más anotaciones por proteína de manera específica, precisa y muy similar.
Es decir, el método recapitula la información obtenida por métodos tradicionales y además anota todo el genoma.
Para responder a la segunda gran pregunta, extrajimos experimentos de transcriptómica publicados para todos los organismos modelo, de los que obtuvimos los genes diferencialmente expresados en distintas condiciones. Calculamos experimentos de enriquecimiento de funciones en esos experimentos de RNAseq para extraer términos enriquecidos. Seguidamente reemplazamos las anotaciones de referencia por las predicciones realizadas por ProtTrans, y repetimos el proceso anteriormente descrito. Con las anotaciones y predicciones, calculamos la similitud semántica entre términos enriquecidos a partir de anotaciones reales versus anotaciones predichas obteniendo valores altos, indicativos de una gran similitud.
En otras palabras, las anotaciones predichas capturan la misma información funcional, lo que implica que estos métodos sirven para abordar experimentos asociados a identificación de funciones en organismos no modelo. Todos estos resultados se han publicado en Barrios-Núñez et al., en 2024. Visto el rendimiento y la fiabilidad de este método, decidimos
usarlo a gran escala, respondiendo a nuestra última pregunta, y para ello se anotaron unos 24 millones de genes de 1.000 especies de animales basales, invertebrados, muy relevantes para estudios de biodiversidad en procesos de terrestrialización. Los métodos tradicionales de referencia anotaban únicamente alrededor de un 50% de los proteomas analizados, dejando en torno a un 50% de media sin anotar, lo que denominamos el “proteoma oscuro”.
Los modelos de lenguaje permitieron anotar prácticamente todos los genes de todos los genomas. Cuando analizamos las funciones del proteoma oscuro, observamos enriquecimientos de funciones muy coherentes con la biología de esos organismos. Por ejemplo, en tardígrados, que son unos organismos prácticamente indestructibles, se identificaron genes previamente sin anotar, con funciones asociadas a su indestructibilidad, como términos asociados a resistencia a UV, radiación, calor, etc. Los resultados están accesibles en Martínez-Redondo et al., en un preprint 2024. Para facilitar a la comunidad el acceso a una herramienta que permita anotar proteomas de manera masiva, hemos desarrollado FANTASIA.
En el último congreso de ISMB2024, se presentó en el panel Function-COSI (Community of Special Interest) el resultado preliminar de la competición en predicción de función CAFA5, donde nueve de los diez mejores métodos usaron modelos de lenguaje.
En conclusión, los modelos de lenguaje de proteínas han llegado para quedarse. Son herramientas muy útiles y alternativas a métodos tradicionales por su capacidad de anotar y por su independencia a criterios evolutivos. Son muy rápidos y no requieren de grandes necesidades computacionales.

Para leer más
Barrios-Núñez I, et al. “Decoding functional proteome information in model organisms using protein language models”. NAR Genomics and Bioinformatics 6 (2024) lqae078. https://doi.org/10.1093/nargab/ lqae078
*CAFA (Critical Assessment of Functional Annotation) es un consorcio de científicos expertos en predicción de función que monitorizan el rendimiento de los métodos. Los resultados de estas actividades suelen presentarse en la mayor conferencia de Biología Computatcional (ICSB), en la sesión de la comunidad de interés COSI-Function.
Elnaggar A, et al. “ProtTrans: Toward Understanding the Language of Life Through Self-Supervised Learning” en IEEE Transactions on Pattern Analysis and Machine Intelligence, vol. 44, no. 10, pp. 71127127 (2022).
FANTASIA (https://github.com/MetazoaPhylogenomicsLab/FANTASIA).
Littmann M, et al. “Embeddings from deep learning transfer GO annotations beyond homology”. Science Reports 11 (2021) 1160. https://doi.org/10.1038/s41598-020-80786-0
Martínez-Redondo IG, et al. “lluminating the functional landscape of the dark proteome across the Animal Tree of Life through natural language processing models” bioRxiv (2024). https://doi. org/10.1101/2024.02.28.582465
Nehrt NL, et al. “Testing the ortholog conjecture with comparative functional genomic data from mammals”. PLoS Computational Biology 7 (2011) e1002073. https://doi.org/10.1371/journal.pcbi.1002073
Stoeger T, et al. “Large-scale investigation of the reasons why potentially important genes are ignored”. Plos Biology 16 (2018) e2006643. https://doi.org/10.1371/journal.pbio.2006643
Theißen G. “Orthology: Secret life of genes”. Nature 415 (2002) 741. https://doi.org/10.1038/415741a
IZASKUN LACUNZA
Directora
general
de la FECYT

« En FECYT tenemos la gran responsabilidad de colaborar para que la sociedad participe de los procesos científicos y tecnológicos
Ismael Gaona Pérez / SEBBM
Una pregunta muy general: ¿Qué tan permeable es la ciencia española hacia la sociedad en general?
Si te refieres a si la ciencia está abierta a la sociedad, creo que ha habido una evolución. Durante mucho tiempo, parecía predominar en la comunidad científica la idea de que la ciencia se desarrollaba aislada, impermeable a las preocupaciones de la sociedad, la búsqueda intelectual era el único fin y solo debían rendirse cuentas a los pares. Afortunadamente, esa visión es cada vez menos común. El riesgo de la torre de marfil es claro: desafección ciudadana, incomprensión, y una ciencia desconectada de las necesidades sociales que puede incluso facilitar movimientos anticientíficos.
Desde el año 2002 y hasta la fecha, la FECYT ha analizado de forma pormenorizada la percepción de la población española hacia la ciencia. ¿Cómo ha evolucionado esta percepción? ¿Cómo participa la población en la ciencia y cuál es su interés hacia ella?
En este momento estamos elaborando la encuesta de este año, cuyos datos publicaremos a principios de 2025. En la edición anterior, cuyos resultados publicamos en 2023, el 12,3% de la población entrevistada decía de manera espontánea que les interesaba la ciencia. Además, había un porcentaje muy elevado de personas que citaban la salud (19,8%) y el medio ambiente y ecología (12,6%), temas que también son ciencia.
También hemos detectado una creciente participación en actividades relacionadas con la ciencia y la tecnología, tanto en iniciativas de divulgación y comunicación, como en acciones políticas sobre la ciencia. Entre los encuestados, el 28,1% reconoce haber firmado peticiones o haber participado en manifestaciones en el último año sobre temas relacionados con la ciencia y la tecnología; por ejemplo, la energía nuclear, la biotecnología, el medio ambiente o el cambio climático. Y 1 de cada 10, aproximadamente, afirma haber asistido a reuniones o debates sobre ciencia y tecnología. Esta creciente participación e interés nos señalan que el diálogo entre ciencia y sociedad está más vivo que nunca.
Con motivo del 20 aniversario de la realización de la Encuesta de Percepción Social de la Ciencia, realizamos un análisis de la evolución de algunas de las actitudes hacia la ciencia de la población española durante estos años. A lo largo de este periodo, ha habido un aumento creciente en el interés por la ciencia
y la tecnología, y la sensación de estar informado sobre estos temas. También ha aumentado el número de personas que realiza actividades como la visita a museos de ciencia.
Sin embargo, existen brechas por sexo, edad y nivel de estudios que se mantienen a lo largo de este tiempo, lo que muestra la necesidad de que la comunicación científica sea mucho más inclusiva. La percepción de que los beneficios de la ciencia y la tecnología son mayores que sus riesgos también han aumentado a lo largo del periodo, así como el número de personas que opina que los conocimientos científicos son la mejor base para elaborar leyes y regulaciones y que los ciudadanos deberían tener un papel más importante en las decisiones sobre ciencia y tecnología que les afectan directamente.
Como en muchos otros ámbitos, la COVID-19 ha cambiado la percepción de la ciencia en todo el mundo. Según un estudio de la FECYT, el 78% de los españoles considera que la ciencia y la tecnología han sido fundamentales durante la crisis sanitaria. ¿Cómo ha influido este dato en las prioridades de la FECYT? Durante la pandemia, pudimos comprobar que la sociedad demandaba información rigurosa, bien contrastada y ágil sobre el nuevo coronavirus y las medidas sanitarias para combatir sus efectos. Lo vimos claramente en el crecimiento que experimentó de 2020 a 2022 la audiencia de nuestra plataforma de noticias científicas SINC (agenciasinc.es). Conscientes de esta necesidad, en marzo de 2022 FECYT lanzó un nuevo proyecto, el Science Media Centre España, una oficina de información dedicada a generar recursos y evidencias para dar apoyo a los periodistas en la cobertura de los temas sobre ciencia, salud, tecnología y medioambiente, especialmente aquellos que generan cierta controversia social.
El Centro Común de Investigación de la Unión Europea (JRC), desde 2020, está impulsando iniciativas para fortalecer y conectar ciencia, políticas públicas y sociedad en los países europeos.
¿Qué papel tiene la FECYT en el fortalecimiento de la conexión ciencia-política-sociedad en España? ¿Qué iniciativas ha puesto en marcha FECYT para hacer realidad el asesoramiento científico en las políticas públicas?
FECYT desempeña un papel crucial en el fortalecimiento de la conexión entre la ciencia, la política y la sociedad en España. Nuestro objetivo es facilitar el intercambio de conocimiento y asegurar que la
evidencia científica informe las decisiones políticas, lo que es esencial para abordar los retos actuales.
Una de nuestras principales iniciativas es la gestión de la Oficina de Ciencia y Tecnología del Congreso de los Diputados, la Oficina C (https://oficinac.es). Esta oficina proporciona a los legisladores informes detallados sobre temas de gran relevancia, como incendios, prevención del suicidio, desinformación y envejecimiento. Al ofrecer el conocimiento experto de una manera accesible, apoyamos a los responsables políticos en su tarea parlamentaria. Además, recientemente, hemos lanzado la Convocatoria I+P, que financia proyectos innovadores que utilizan evidencias científicas para desarrollar políticas públicas más efectivas. Con un presupuesto total de dos millones de euros, esta convocatoria apoya el asesoramiento científico y fomenta la colaboración entre la comunidad científica y la administración pública.
A través de estas y otras iniciativas, FECYT trabaja para construir puentes entre la ciencia y la política, promoviendo un ecosistema en el que el conocimiento científico sea un pilar en la formulación de políticas que beneficien a la sociedad.
Los tiempos cambian y los retos en la comunicación científica no son los mismos que, por ejemplo, hace cinco años. Ahora estamos mirando de cerca la inteligencia artificial, el Open Science, etc. ¿Cuáles son los principales desafíos que enfrenta la FECYT en la promoción de la cultura científica en España?
Tenemos la gran responsabilidad de colaborar para que la sociedad participe de los procesos científicos y tecnológicos y, por tanto, el reto es que la comunicación de la ciencia llegue a públicos diversos y sea cada vez más inclusiva.
Hace más de 20 años que gestionamos la Convocatoria de Ayudas para el Fomento de la Cultura Científica y de la Innovación, una convocatoria de ayudas para que organizaciones de todo tipo, públicas y privadas, pongan en marcha acciones de comunicación de la ciencia dirigidas al público en general. Pero nuestro objetivo es estimular el sentido crítico y la toma informada de decisiones, y por eso estamos trabajando en otra dimensión que queremos añadir a esa conexión entre ciencia y sociedad: las políticas públicas y queremos tener en cuenta un nuevo grupo de interés: los decisores públicos. Creemos que la ciencia debe ser un factor tenido en cuenta a la hora de diseñar políticas públicas, es decir, que las políticas públicas estén informadas por la evidencia científica. Ya hemos abierto algunas líneas de trabajo en esa área, como mencionaba antes la Oficina C o la convocatoria de Innovación en Políticas públicas. Estamos aprovechando la experiencia que FECYT tiene, y aspiramos a fomentar un ecosistema e impulsar
un cambio cultural de la misma manera que hemos hecho en Comunicación social de la ciencia. Y todo ello encaja con otra de nuestras líneas de actuación más relevantes: la ciencia abierta. Queremos que el conocimiento sea un bien público sostenible, accesible, inclusivo y multilingüe, libre de sesgos y abierto a la sociedad. Damos soporte al Ministerio de Ciencia, Innovación y Universidades en la puesta en marcha de la Estrategia Nacional de Ciencia Abierta (ENCA). Los servicios de FECYT hacen accesibles los resultados de la investigación financiada con fondos públicos, tanto para la comunidad científica que los produce, como para la sociedad que los financia. Con ello potenciamos la reproducibilidad de la ciencia y la reutilización de los resultados.
El concepto de ciencia ciudadana está ganando relevancia a nivel mundial. ¿Qué importancia tiene este enfoque para la FECYT y cómo está promoviendo la participación ciudadana en proyectos de investigación que involucran a la sociedad?
La ciencia ciudadana en España —que cada vez es más diversa en cuanto a temas, metodologías, alcance e inclusividad— genera un impacto social, medioambiental y educativo relevante, además de científico. Creemos que la ciencia ciudadana promueve el acceso en abierto, universal, al conocimiento, a la ciencia. Crea cambio y transformación en la sociedad; promueve la innovación tecnológica y social, además de favorecer la orientación de fondos públicos para asuntos de interés y preocupación social. También promueve las políticas en un sentido bottom-up, de la sociedad a las instituciones.
Aunque FECYT en su Convocatoria de ayudas para el fomento de la cultura científica y de la innovación incluía la ciencia ciudadana entre sus objetivos desde el año 2013, fue a partir de 2018 cuando incluyó una línea de actuación específica para proyectos de ciencia ciudadana.
En 2022, FECYT lanzó esta misma Convocatoria en la que se incluía de manera excepcional una categoría singular de proyectos de ciencia ciudadana en coordinación, financiada por el Ministerio de Universidades con una dotación de 400.000 €. Además, se incluía como parte de la categoría general de Proyectos de comunicación social de la ciencia y la tecnología (dotada con más de 2.000.000 €) un objetivo específico de “Estimular la participación ciudadana en la ciencia y la tecnología” al que podían acogerse los proyectos de ciencia ciudadana que no cumpliesen los requisitos de la categoría singular. En 2023 y 2024 esta categoría y objetivo específico se han mantenido. El resultado es que cerca de 229 proyectos han sido presentados desde la convocatoria de 2018 en la categoría singular de ciencia ciudadana y más de 1.300.000 euros han sido dedicados a promover e

impulsar específicamente la participación ciudadana en proyectos de investigación.
Uno de los retos más importantes en la divulgación científica es comunicar información compleja de manera accesible. ¿Qué estrategias está adoptando la FECYT para hacer la ciencia más comprensible y atractiva para el público general? ¿Qué formatos son los que más llegan al público?
Según la última Encuesta de Percepción Social de la Ciencia y la Tecnología publicada en 2022, la realización de actividades relacionadas con la ciencia y la tecnología se ha incrementado respecto a los últimos años. La actividad que se ha realizado de manera más habitual es hablar de ciencia e investigación con familiares y amigos (61,4%) y ver o escuchar programas de televisión o radio sobre ciencia (57,4%). Estos resultados ponen en relieve que las acciones en redes sociales, los grandes proyectos audiovisuales o la ciencia sobre los escenarios (monólogos, improvisación, teatro…) viven ahora un buen momento al ser una de las opciones más atractivas a la hora de realizar actividades de divulgación científica. Solo de Ciencia, un certamen iberoamericano de monólogos científicos, que cumple ya su segunda edición, ha sido un éxito al involucrar activamente a
los investigadores en la comunicación de la ciencia en un formato diferente. También Iniciativas que interrelacionan el arte, la ciencia, la tecnología y la sociedad (ACTS), la ciencia ciudadana o la educación STEAM, entre otros, también han reconfigurado el imaginario social en las dos últimas décadas, abriendo nuevos formatos de proyectos que reconfiguran la participación, habitualmente pasiva, de la sociedad en las actividades de divulgación científica. Estas prácticas ocurren en espacios artísticos, laboratorios de creación y ciudadanos, etc., lugares donde es posible investigar y utilizar metodologías diferentes a las de los lugares de producción de conocimiento tradicionales. Todas estas iniciativas vienen siendo apoyadas por FECYT en los últimos años a través de líneas de financiación específicas, proyectos propios y en colaboración, con el objetivo de entender mejor la complejidad de las cuestiones contemporáneas (por ejemplo, el cambio climático y nuestra relación con el medioambiente, el impacto de la tecnología y los avances científicos, etc.) y buscar soluciones a los problemas y promover políticas frente a los mismos, favoreciendo los enfoques inter/transdiciplinares y abiertos, en los que la ciudadanía pueda implicarse activamente.
Recientemente, la FECYT ha lanzado varias iniciativas y convocatorias (I+P, Cultura Científica, REBECA Practice...) para el emprendimiento, para acercar la ciencia a la sociedad o para el colectivo investigador. ¿Podría destacar alguna que haya tenido un impacto significativo, teniendo en cuenta que la participación en actividades científicas se ha duplicado en los últimos cinco años, según sus informes?
La Convocatoria I+P es quizás la más reciente y ha sido una satisfacción comprobar el gran interés que ha suscitado. Hemos recibido 370 solicitudes de 127 entidades. Su objetivo es impulsar proyectos que fomenten la innovación pública y el asesoramiento científico, con un enfoque basado en la colaboración entre las administraciones públicas y la comunidad científica. Con esta convocatoria buscamos fomentar una cultura de innovación en las administraciones públicas, para que la evidencia científica sea un pilar fundamental para las políticas públicas. Pero, sobre todo, queremos impulsar una transformación cultural y crear un ecosistema, lo mismo que hemos venido haciendo con la Convocatoria de Ayudas para el fomento de la Cultura científica y de la Innovación. Esta convocatoria no es precisamente nueva, pero no quiero dejar de mencionar su impacto. Gracias a su financiación hemos impulsado nuevos formatos de la ciencia y líneas de comunicación social de la ciencia como la ciencia ciudadana o las iniciativas de arte, ciencia, tecnología y sociedad; Durante 18 años hemos financiado 3.800 proyectos que han permitido la participación de cerca de 36 millones de personas en todo el territorio y hemos logrado consolidar una red de Unidades de Cultura Científica de más de 120 nodos distribuidos por todo el país. Volviendo a iniciativas más recientes, pusimos en marcha el programa de mentorazgo REBECA (REsearchers BEyond aCAdemia Mentoring Programme, en inglés), que busca ofrecer nuevas oportunidades laborales más allá de la investigación académica al personal investigador que se encuentra en las primeras etapas de su desarrollo profesional. A lo largo de las diferentes ediciones, hemos puesto en marcha cientos de parejas de mentores e investigadores que han permitido al personal investigador participante conocer a profesionales con formación científica que han desarrollado su carrera profesional en otros ámbitos más allá de la investigación pública. Este acercamiento les ayuda a reflexionar sobre sus propias competencias y opciones profesionales y amplía su mirada. Los niveles de satisfacción y aprendizaje de todos los participantes en el programa demuestran su eficacia.
En este contexto, lanzamos la herramienta REBECA Practice que ha tenido un gran impacto porque permite a los investigadores explorar diferentes puestos relacionados con la investigación y la innovación que
se desarrollan en el sector público y privado. Aunque el mercado laboral demanda las competencias y alta cualificación que el personal investigador adquiere en su formación, todavía queda mucho por hacer para facilitar la movilidad intersectorial. Por eso es necesario formar, poner en valor y profesionalizar opciones de carrera en ciencia. La herramienta REBECA Practice permite, mediante un caso simulado, acercar otras profesiones como periodista científico, técnico de evidencia científica o gestor de la innovación.
¿Qué papel juega la FECYT en la lucha contra la desinformación y cómo se están implementando estrategias para corregir esta tendencia, y mejorar la educación científica en la población?
La desinformación es un fenómeno complejo en el que intervienen numerosos actores y dinámicas sociales y políticas. Destacaría el papel que juega FECYT en la participación en proyectos europeos como Iberifier, que es un observatorio de medios digitales de España y Portugal, impulsado por la Comisión Europea donde se analiza el ecosistema ibérico de los medios digitales y hace frente al problema de la desinformación. En 2022 elaboramos un informe analizando esas dinámicas en el marco del proyecto. Más recientemente hemos elaborado un informe en la Oficina C, la Oficina de Ciencia y Tecnología del Congreso de los Diputados. Ambos informes señalan cómo la tecnología ha favorecido una amplificación sin precedentes de la desinformación y sus efectos, convirtiéndola en una desatacada amenaza para los sistemas democráticos. Los expertos que participan en el informe destacan la importancia de la alfabetización mediática, por ejemplo, para identificar las fuentes que son fiables. Es indudable el papel central que el periodismo y los profesionales de la información tienen para combatirla. De ahí nuestros esfuerzos en impulsarlo desde SINC, una plataforma de actualidad científica que produce contenidos periodísticos sobre ciencia, rigurosos y atractivos para el público general, con licencia Creative Commons. También hemos puesto en marcha el Science Media Centre España para que la ciudadanía esté informada de los temas científicos que generen controversia y mejorar la calidad del debate público. Para ello pone a disposición de los periodistas recursos que les permiten elaborar buenos contenidos de ciencia en los plazos de tiempo que marca la actualidad, como reacciones de voces expertas, documentos de evidencias e información de contexto.
Además, en nuestro proyecto Ciencia de la Comunicación Científica trabajamos para analizar la comunicación pública de la ciencia, identificar necesidades y promover acciones destinadas a impulsar una comunicación social de la ciencia más eficaz, ética y profesional.

LA IMPORTANCIA DE ELEGIR
EL MODELO CELULAR ADECUADO
Líneas celulares y tejidos adaptados a tus necesidades.
Los incesantes avances en técnicas de análisis celular y molecular han permitido en los últimos años un aceleración del descubrimiento científico en todas las áreas de la investigación biológica Sin embargo, de poco sirven las más revolucionarias y sofisticadas tecnologías, si no se parte de un modelo de estudio adecuado
Frente a los organismos vivos multicelulares y complejos, los tejidos ex vivo y las líneas celulares se han convertido en una herramienta crucial en investigación, debido a su relativa sencillez y facilidad de mantenimiento en el laboratorio. Pero conseguir un modelo celular simplificado, a la par que representativo, no siempre es tan fácil.
En Condalab, empresa líder en soluciones de laboratorio con más de 60 años de experiencia, apostamos por herramientas y servicios que puedan permitirte alcanzar el modelo que verdaderamente se ajusta a las exigencias de tu investigación.
Líneas inmortales para mayor reproducibilidad.
Olvídate de tener que reponer tus primarias continuamente y la variabilidad de resultados que conlleva. Contamos con la mayor colección de líneas inmortales de diversos sistemas y especies. ¿Te interesa inmortalizar las primarias de tus pacientes o de tus modelos animales para usarlas a largo plazo? Contrata el servicio completo o solicita nuestros virus empaquetados ready-to-use.
Modificaciones estables a la carta sin complicaciones.
¿Necesitas generar un knockout por CRISPR? ¿Sobreexpresar o silenciar tu gen de interés?
Ahorra tiempo y frustraciones: consulta nuestra biblioteca prediseñada de sistemas para expresar, editar o silenciar cualquier gen de humano, rata o ratón. Si no encuentras lo que buscas, podemos customizar el producto o servicio a tu medida.
Tejidos frescos o congelados; modelos más representativos.
A medio camino entre las líneas celulares y los organismos vivos, las fracciones celulares y tejidos son un gran aliado en farmacología y cosmética, ya que permiten hacer pruebas fiables de toxicología o eficacia con diversos reactivos.
Consulta nuestra colección de láminas de piel, silensomas de hígado, fracciones de sangre y plasma o tejido pulmonar.
¿Quieres saber más? Contacta con nosotros: elena moreno@condalab com +34 91 761 02 00 / www.condalab.com

VOCACIONES: LA CRISIS SILENCIOSA
IGP / SEBBM
La ciencia, la innovación y el emprendimiento necesitan de materia gris y, sobre todo, de vocaciones que nos aseguren respuestas en forma de talento profesional para atender las grandes macrotendencias del siglo XXI, como las transiciones energética y digital. Se estima, por ejemplo, que la industria biotecnológica en España generará 11.000 empleos en los próximos cinco años. La cuestión es si contamos con recursos suficientes para atender esta demanda y en qué condiciones.
¿Quién no recuerda aquella frase de Mamá, quiero ser artista popularizada por Concha Velasco?
Esta expresión cautivó el deseo de toda una generación por brillar en los escenarios y conquistar el mundo del espectáculo. Ser artista representaba, y así lo sigue siendo, una mezcla de talento, dedicación y ambición por triunfar frente a un público real. Sin embargo, hoy esa frase ha evolucionado y en pleno siglo XXI, podemos decir sin tapujos algo así como Mamá, quiero ser youtuber, o Quiero ser influencer
Esta transformación no es casual; refleja cambios profundos en las aspiraciones de un segmento de la población que mide el éxito en likes. El estudio La generación Z desea ser youtuber y/o influencer. Factores que determinan esta tendencia publicado en 2022 por María Pilar Arenas, Antonia RamírezGarcía y Rosa Ruiz (Universidad de Córdoba, España) en Comunicación y Tecnologías emergentes revela que el 46,9% de los adolescentes siente atracción por las profesiones de youtuber y/o influencer, lo que contrasta con el perfil profesional aspiracional de hace apenas dos décadas, cuando carreras como medicina, derecho o ingeniería figuraban entre las más deseadas.
Parte de este cambio en las aspiraciones juveniles puede atribuirse a la revolución digital. Según un
estudio de la American Psychological Association, la constante exposición pública fomenta una percepción del éxito como algo superficial, todo lo contrario a lo que ocurre con la construcción de lo que llamamos vocaciones científicas, que necesitan cimientos sólidos tanto culturales como educativos para que puedan asentarse en un terreno tan dinámico y versátil como es la innovación, la investigación o el emprendimiento.
Aunque su herencia sigue viva, lejos quedan referentes científicos que abrían camino en el mundo y servían de inspiración para jóvenes que veían en la ciencia una opción de vida. Mientras Concha Velasco encandilaba al público y a la crítica, científicos como Severo Ochoa, premio Nobel de Fisiología o Medicina en 1959, mostraban al mundo que el talento español podía alcanzar la cima de la excelencia; la bioquímica Margarita Salas marcaba un antes y un después en la ciencia española, y se convirtió en un símbolo del esfuerzo y la dedicación necesarios para abrirse paso en un campo dominado por hombres…
Dudas razonables
Esta transformación en los modelos de éxito, en la búsqueda de referentes, plantea retos importantes. España viene arrastrando desde hace años una crisis silenciosa que lanza serias dudas sobre cómo de

preparado está nuestro país en cuestión de talento, de personas o de recursos para afrontar las grandes megatendencias del siglo XXI, como son la transición energética o la digitalización. Y lo cierto es que, según la radiografía que nos facilitan instituciones, organismos internacionales, fundaciones o empresas privadas, no pinta muy bien el asunto.
Vaya por delante el primer dato global extraído del informe The future of jobs 2020 del World Economic Forum (https://www.weforum.org/publications/thefuture-of-jobs-report-2020/), que prevé que unos 85 millones de puestos de trabajo se volatilizarán como consecuencia de la irrupción de la digitalización, por una tasa de reposición de 97 millones de nuevos empleos relacionados con la inteligencia artificial o la creación de algoritmos, entre otros. Es decir: la demanda de talento no quedaría cubierta de seguir la tendencia abierta desde hace décadas por la desafección del alumnado por las disciplinas STEM (Ciencia, Tecnología, Ingeniería y Matemáticas), imprescindibles para el desarrollo social y económico de la sociedad tal y como la conocemos en la actualidad. Y es que este mismo documento de análisis proyecta que para el año 2030, el 75% de los empleos más demandados requerirán competencias en STEM, lo que pone de relieve la urgencia de incrementar la formación en estas áreas.
La Fundación CYD es explícita al respecto (https:// www.fundacioncyd.org/informe-cyd-2023-que-titulaciones-estudian-los-universitarios-en-espana-y-como-es-su-insercion-laboral/) y nos alerta del reducido porcentaje de titulados en STEM en nuestro país, cuarto lugar por la cola entre los 27 países de la UE, tras Chipre, Malta y Bélgica; y pese a que los países que invierten en programas sólidos de educación STEM desde etapas tempranas son los que logran no solo atraer más estudiantes a estas disciplinas, sino también retener a profesionales cualificados en áreas de alta tecnología, según Eurostat (2022), solo el 6% de los jóvenes españoles completan estudios en áreas STEM, frente al 9% de la media europea.
De forma adicional, el estudio de Fundación COTEC e IVIE Mapa de Talento 2023 (https://cotec. es/informes/metricas-mapa-del-talento-autonomico/) apuntala a España como un país exportador de conocimiento: el 43% de los trabajadores españoles que emigraron en 2019 lo hicieron para ocupar un puesto altamente cualificado, mientras que sólo el 11,3% de los extranjeros que vinieron a trabajar a España ocupaban ese perfil. Además, el 39,9% de los emigrantes españoles acabaron desempeñando ocupaciones elementales o manuales, frente al 84,9% de los extranjeros que llegaron a España; sin
POLÍTICA CIENTÍFICA
olvidar que más de la mitad de la masa de emigrantes que se fueron tenía estudios superiores, frente a casi la cuarta parte de los inmigrantes que llegaron. Una tormenta perfecta.
Esta situación ya se siente en las empresas, que se muestran abrumadas ante esta situación deficitaria. Por ejemplo, Fundación Telefónica estima que la demanda de profesionales TIC crecerá un 17% en los próximos años, mientras que, en otro escenario, el sector biotecnológico proyecta un crecimiento anual del 7,4% hasta 2030, prediciendo esta comentada necesidad de personal investigador y técnico. Así, se estima que la industria biotecnológica en España generará 11.000 empleos en los próximos cinco años (ASEBIO), con un volumen de mercado de unos 6.000 millones de euros para 2025.
Un informe realizado por Ramstad para 2024 (entre 800 empresas) revela que conseguir el talento que necesitan es el “principal desafío en materia de recursos humanos”. Un 75% de las empresas experimenta de forma directa la falta de profesionales cualificados, seguido de la competencia en el sector (es decir, que el profesional tiene muchas empresas entre las que elegir).
Pero vamos al origen
La ministra de Ciencia, Innovación y Universidades, Diana Morant, lanzó hace meses una interesante reflexión en un foro organizado por Europa Press sobre la formación y capacitación del profesorado español, primer pilar del despertar científico del alumnado. “Muchos profesores y profesoras ya vienen con sus propias manías hacia las Matemáticas, con su propia pelea con las carreras de Ciencias, lo que terminan transmitiendo también a los alumnos y alumnas”, aseguró la ministra. “Existe un sesgo en la carrera de Magisterio, que es cursada mayoritariamente por mujeres, y como tales, a menudo transmitimos esos mensajes sociales a los estudiantes. Otro dato importante es que la mayoría de los profesores y profesoras han elegido previamente carreras de letras, no de ciencias, por lo que ya traen consigo sus propios prejuicios hacia las Matemáticas, su propia pelea con las carreras científicas, y eso lo transmiten. Es necesario trabajar este tema con el profesorado para que pierda el miedo a las disciplinas científicas, evitando así que lo transmitan, y ofrecerles una formación específica con perspectiva de género”. (https://youtu. be/JS0Y6FJxI74?feature=shared).

Las palabras de Morant vienen al pelo para intentar justificar los resultados del último Informe del Programa para la Evaluación Integral de Alumnos (PISA 2022) que elabora cada tres años la Organización para la Cooperación y el Desarrollo Económico (OCDE), y descargar, en parte, la responsabilidad en el cuerpo docente. En la evaluación realizada en Matemáticas los estudiantes españoles obtuvieron 473 puntos, lo que supone su peor resultado en la historia del informe (ya que desde 2003 siempre han obtenido al menos 480 puntos) y 8 puntos menos que en 2018, cuando obtuvieron 481 puntos.
Para Patricia Senent, Graduada en Bioquímica por la Universidad de Murcia y profesora de Educación Secundaria en Biología y Geología (además de youtuber con canal propio), hay varios factores que pueden estar relacionados con estas deficiencias. “Para empezar, la manera en que el sistema educativo estructura las asignaturas científicas. En muchas materias de Ciencia, como Física o Química, los contenidos a impartir son muchísimos, además de ser muy abstractos y matemáticos, y se presentan como un montón de teorías y fórmulas que pueden ser difíciles de entender. Eso puede lograr que los estudiantes se sientan desmotivados o incapaces de comprender ciertos temas”.
Por otro lado, sugiere la docente, “la mayoría de las clases se centran en la teoría, porque no tenemos tiempo ni recursos para realizar actividades prácticas”. “A pesar de los esfuerzos que realizamos para hacer la Ciencia más amena, la cantidad de contenidos a impartir, la falta de materiales (trabajo en un instituto público) y las pocas horas semanales asignadas a cada asignatura nos obligan a priorizar la teoría sobre la práctica. Esto nos limita y, aunque intentamos incluir ejercicios divertidos y prácticas experimentales, muchas veces vamos a contrarreloj porque hay que terminar el temario como sea”. ¿El resultado? Los estudiantes, según manifiesta, perciben la Ciencia como una serie de conceptos que deben memorizar, en lugar de una asignatura práctica y dinámica que tiene aplicaciones en el mundo real. “Habría que centrarse también en una reestructuración del temario para permitir un enfoque más práctico y así mejorar la motivación y el interés del alumnado”, declaró. Daniela es una estudiante sevillana de Bachillerato. Reconoce que la Investigación, la Ciencia y la Innovación son conceptos esenciales de avance social y generadores de bienestar y riqueza. Sin embargo, y pese a sus tímidos flirteos con asignaturas de Ciencias, se ha decantado por una carrera de Letras. “Veo bastante lejos el mundo de los laboratorios, de la docencia, y lo veo desde la perspectiva de tiempo, porque ejercer la carrera investigadora es bastante complicado”, subraya.
Como Daniela, en España, el Informe sobre la Percepción Social de la Ciencia y la Tecnología de 2022 (FECYT) revela que el 77% de la población tiene una visión positiva de la Ciencia, pero solo el 12% de los jóvenes se plantea cursar estudios relacionados con disciplinas STEM, datos que muestran una tendencia preocupante porque, aunque la ciencia es valorada como “importante”, los jóvenes no logran conectar esta apreciación con una decisión profesional o vocacional.
Profesorado: la falta de consenso, hándicap principal Pero además de la opinión de Patricia Senent, también existen encuestas dirigidas al profesorado. Y el estudio VII Informe Young Business Talents: La visión del profesor va más allá y destaca los puntos negros sobre la situación educativa en España. Por ejemplo, las conclusiones de este trabajo señalan que la ausencia de un pacto de Estado educativo es identificada por el 40,9% del profesorado como la principal debilidad, destacando, además, la falta de consenso en los cambios educativos.
En segundo lugar, señalan la alta ratio de alumnos por clase y la falta de recursos educativos, con un 17,3% y un 16% de los profesores, respectivamente. Con relación a la preparación para la vida laboral, el 73,7% de los profesores considera que los alumnos no están adecuadamente preparados, siendo la falta de participación de los alumnos y sus familias (67%) y la saturación en las aulas (33%) las principales causas identificadas.
Abordar la brecha de género en STEM
Además de la falta generalizada de vocaciones STEM, España se enfrenta a otro reto significativo: la brecha de género. Aunque en nuestro país las mujeres se incorporan cada vez más y a mayor ritmo que los hombres a las carreras científicas, no lo hacen por igual en todas las áreas, según el informe Científicas en Cifras 2021. Este destaca la necesidad de fomentar las vocaciones científicas y técnicas entre las jóvenes, como demuestra el hecho de que, aunque las mujeres son el 56% de las universitarias, en áreas como la ingeniería o la tecnología solo representan el 25,4%.
Según CYD 2023, esta subrepresentación limita el acceso de las mujeres a sectores de alto crecimiento y perpetúa estereotipos de género que disuaden a las niñas de elegir carreras científicas o tecnológicas. Esta brecha de género no es solo un problema de equidad y tiene profundas repercusiones para la economía y la innovación. La OCDE (2019) ha demostrado que los equipos diversos, tanto en género como en experiencia, tienden a ser más innovadores y mejores a la hora de resolver problemas complejos. La inclusión de más mujeres en STEM no solo es una
POLÍTICA CIENTÍFICA
cuestión de justicia social, sino también de mejorar la competitividad y la capacidad innovadora de las empresas y las industrias.
Por otro lado, y como buena noticia, es alentador observar que España sobresale en el ámbito de las patentes, con un 46% de las solicitudes que incluyen al menos a una mujer inventora, superando significativamente el promedio del 27% de los Estados miembros de la Oficina Europea de Patentes (OEP). Sin embargo, es importante destacar que la representación de mujeres varía según el sector. Por ejemplo, en ingeniería mecánica, solo el 14% de los inventores son mujeres, mientras que en química esta cifra alcanza el 50%.
Periodismo científico y divulgación
La encuesta on-line anual Edelman Trust Barometer 2024, realizada en 28 países y que ha contado con la participación de 32.000 personas, formula una pregunta muy directa en su último sondeo: ¿En quiénes confiamos para comunicar honestamente la innovación? La respuesta no es baladí: Los científicos, con un 82% de las afirmaciones, mientras que los periodistas ocupan la sexta plaza (36%) por detrás de “alguien como yo” (74%), expertos técnicos en empresas (70%), representantes de ONG’s (44%) y CEO’s (43%). (https://www.edelman.com.es/sites/g/ files/aatuss396/files/2024-03/2024%20Edelman%20 Trust%20Barometer_Spain%20Report.pdf).
Patricia Senent es, precisamente, exponente de un movimiento que tiene en el acercamiento de la Ciencia a la Sociedad su objetivo, dando así respuesta a uno de los factores más subestimados que influyen en la elección de carreras científicas entre los jóvenes: la divulgación científica y el acceso a información científica adaptada a nuevos formatos digitales y rigurosa. “Me dedico a inspirar y educar tanto en el aula como en plataformas digitales. Como profesora de secundaria, me esfuerzo por fomentar la vocación científica entre mis estudiantes. El año pasado, en el instituto, llevamos a cabo un proyecto de pódcast llamado “Mujeres en la Ciencia”, donde las alumnas entrevistaban a profesoras de carreras científicas para conocer sus experiencias y desafíos, y así promover la igualdad y mostrar que la Ciencia es para todos”, ha asegurado a la revista de la SEBBM. Plataformas como YouTube, TikTok y Twitch han demostrado ser herramientas poderosas (incluimos los pódcasts) para acercar la ciencia a los jóvenes. Canales de divulgación como QuantumFracture ha conseguido más de 3,5 millones de suscriptores explicando teorías de la relatividad o del universo a través de animaciones visuales que hacen que la ciencia sea comprensible incluso para quienes no tienen una formación especializada.
Precisamente, el creador de QuantumFracture, José Luis Crespo, participa en el programa Digitalizarte (https://programadigitalizarte.com/), un proyecto formativo online impulsado por Google, la Fundación Alternativas y la firma 2btube para dar herramientas a cualquier autor, institución o profesional del ámbito científico o artístico y que les permitan darse a conocer a través de YouTube. El programa está abierto a cualquier creador y tiene una duración de siete semanas.
El papel de la SEBBM
De forma paralela, las sociedades científicas, verdaderas agentes del conocimiento, intentan romper esta desafección entre sociedad y ciencia mediante acciones de naturaleza múltiple: desde ponencias, exposiciones virtuales, juegos interactivos, etc. Desde 2009, la Sociedad Española de Bioquímica y Biología Molecular (SEBBM) cuenta con un comité de divulgación dedicado específicamente a acercar la ciencia a la sociedad, siendo pionera en la creación de actividades orientadas a este propósito. Entre sus iniciativas destacan los artículos de divulgación en las secciones ‘Rincón del aula’ y ‘Acércate a…’ (recopilados ahora en los Cuadernos de Divulgación), la ‘Pinacoteca de la Ciencia’, vídeos en la sección ‘Experimenta con nosotros’, el concurso ‘Dibuja una persona que se dedique a la ciencia y ponle un nombre’ (dirigido a estudiantes de primaria) y los talleres organizados durante la ‘Noche Europea de los Investigadores’, la ‘Semana de la Ciencia’ y el ‘Día Internacional de la Mujer y la Niña en la Ciencia’. Según Sara García Linares, vocal de Jóvenes y Educación de la SEBBM, “estas actividades juegan un doble papel: no solo acercan el mundo científico a los estudiantes más jóvenes, sino que también permiten que el público comprenda y apoye proyectos científicos, fomentando la transparencia y la participación ciudadana”. La divulgación facilita que el conocimiento especializado llegue a la sociedad y a los responsables de la toma de decisiones, promoviendo la creación de políticas informadas, basadas en la evidencia científica.
La FECYT, un actor clave
El documento Green Paper on Citizen Science: Citizen Science for Europe describe la Ciencia Ciudadana como el compromiso del público general en actividades de investigación científica. En 2006, la FECYT comenzó a gestionar la convocatoria para el fomento de la cultura científica y, desde entonces, se han impulsado 3.800 proyectos para acercar la ciencia a la ciudadanía, con una financiación de 67 millones de euros y más de 156 millones movilizados de otras entidades públicas y privadas.
Ayuntamientos, OPIs, universidades y otras
instituciones han liderado muchos de estos proyectos, destacando un alto porcentaje de iniciativas encabezadas por mujeres. Además, más de 36 millones de personas han participado en las actividades impulsadas. Asimismo, se han creado en todo el país 1.200 Unidades de Cultura Científica y de la Innovación (UCC+I), que actúan como intermediarias entre las instituciones que las acogen y los ciudadanos, con el objetivo principal de promocionar la cultura científica, tecnológica y de la innovación a través de actividades de diversa tipología: comunicación científica, divulgación, formación, etc. Estas unidades se han convertido en uno de los principales agentes en la difusión y divulgación de la ciencia y la innovación en España, constituyendo un servicio clave para mejorar e incrementar la formación, la cultura y los conocimientos científicos de los ciudadanos.
El servicio de noticias científicas SINC o el Science Media Center también son ejemplos, según FECYT, “de cómo nutrimos de evidencia científica los procesos de toma de decisiones. Nuevos formatos y líneas de comunicación están haciendo la ciencia más abierta e inclusiva”.
Y, por último, la Semana de la Ciencia y la Noche de los Investigadores, ejemplos de actividades de participación ciudadana. La Fundación Española para la Ciencia y la Tecnología (FECYT) ha financiado con 115.363,14 € a través de su Convocatoria de ayudas para el Fomento de la Cultura Científica de 2023, diversas actividades que diez comunidades autónomas han desarrollado durante el mes de noviembre para conmemorar la Semana de la Ciencia. Las regiones que han recibido financiación para estas iniciativas son: Andalucía, Asturias, Castilla-La Mancha, Castilla y León, Cataluña, Comunidad de Madrid, Comunidad Valenciana, La Rioja, Navarra y la Región de Murcia. Entre las actividades financiadas se encuentran talleres de diferentes temáticas orientados a público general y escolar, conferencias, monólogos científicos, debates, exposiciones, juegos, concursos, catas, visitas guiadas, cine, experimentos, demostraciones y otras muchas propuestas.
ONAC o cómo traducir la Ciencia a los decisores
El Gobierno presentó el pasado mes de junio la Oficina Nacional de Asesoramiento Científico (ONAC), cuyo principal objetivo es que la voz de los científicos sea escuchada de forma integral en las administraciones. De este modo, los 22 ministerios (incluido el de Ciencia, Innovación y Universidades) contarán con un asesor científico con el que mejorar la asesoría en la toma de decisiones políticas. De forma paralela, también se ha puesto en marcha un programa de estancias, con el que varias decenas de científicos atenderán a las peticiones de las distintas carteras

ministeriales en períodos de seis meses. Todos ellos estarán dirigidos por el sociólogo Josep Lobera. Si bien ya existen experiencias de asesoramiento científico, la ONAC trabaja para reforzar el papel del conocimiento científico en la toma de decisiones del Ejecutivo mediante el diseño y la implementación de mecanismos institucionales para el asesoramiento científico a la Administración General del Estado. Uno de estos mecanismos acaba de ser constituido de forma oficial gracias a un acuerdo del Consejo de Ministros para crear el Grupo de Trabajo para el Asesoramiento Científico al Gobierno y una de sus principales misiones ha sido definir el perfil de los asesores científicos que mejor se ajustan a las necesidades de los 22 ministerios, y que se han incorporado recientemente, seleccionados de entre las 1.601 expresiones de interés recibidas. Gracias a su creación, la ONAC sigue avanzando en el cumplimiento de su objetivo fundacional: reforzar el papel del conocimiento científico en la toma de decisiones. “Se trata de un grupo de trabajo similar al que ya poseen otras instituciones, como el SAPEA que la Comisión Europea constituyó en 2016 para apoyar su Mecanismo de Asesoramiento Científico”, señala el director de la ONAC, Josep Lobera.

ALFRED NOBEL
LOS PREMIOS NOBEL DE FÍSICA Y DE QUÍMICA DE 2024
Javier Sancho
Instituto de Biocomputación y Física de Sistemas Complejos
Universidad
de Zaragoza
En una decisión aparentemente coordinada, la Real Academia Sueca de las Ciencias ha concedido este año los Premios Nobel de Física y de Química (Figura 1) al desarrollo y aplicación de procedimientos de análisis masivo de datos basados en redes neuronales artificiales. La estructura y funcionamiento de las redes neuronales, que se inspiró inicialmente en la neurociencia, se ha nutrido de herramientas y conocimientos de la física y ha encontrado extraordinarias aplicaciones en biología estructural y en numerosos aspectos de nuestra vida cotidiana.
Una red neuronal está constituida por nodos interconectados. Cada nodo almacena un número que constituye su valor y se interconecta con otros nodos de la red según valores que describen la fortaleza de cada conexión. La red suele tener varias capas. Cuando un nodo de la capa de entrada recibe un dato, modifica el valor que tiene almacenado y también
los valores de los nodos con los que está conectado. Cuando la red termina de procesar los datos disponibles, los valores que aparecen en los nodos de la capa de respuesta constituyen el resultado de su análisis. Las redes neuronales son entrenadas con una gran cantidad de datos que les permiten optimizar las intensidades de sus conexiones mediante cálculos automatizados. De esta manera, consiguen aprender autónomamente a llevar a cabo a gran velocidad algunas tareas complejas que las personas hacemos bastante bien (a nuestro ritmo) con nuestras propias redes neuronales (p. ej. traducción de textos, interpretación de imágenes, generación de textos o de imágenes realistas). También consiguen realizar tareas fuera de nuestro alcance, como el análisis simultáneo de gran cantidad de datos o el descubrimiento de patrones predictivos sutiles.
Las personas que diseñan una red neuronal albergan la

esperanza de que su arquitectura y reglas de funcionamiento sean las adecuadas para realizar la tarea prevista, pero ignoran habitualmente de qué manera lo van a conseguir (con qué valores de las conexiones entre los nodos). Esos valores “aparecen” durante el entrenamiento. En realidad, las complejas tareas que resuelven las redes neuronales podrían también resolverlas modelos predictivos explícitos que fueran suficientemente precisos y utilizaran únicamente parámetros con significado físico y reglas prefijadas. Tales modelos, nos evitarían algunas de las preocupaciones que nos causa la “Inteligencia artificial” (IA) y que se están comenzando a debatir abiertamente pues, al no ser capaces de aprender nada, no podrían escapar a nuestro control. Es probable que estos modelos deterministas no lleguen pronto, así es que nos toca disfrutar de
las grandes hazañas que realizan las redes neuronales artificiales y abordar sin demora el estudio de los peligros que plantean. «Por los descubrimientos e inventos fundacionales que permiten el aprendizaje automático con redes neuronales artificiales», el Premio Nobel de Física ha sido otorgado este año al estadounidense John J. Hopfield (91 años) y al británico, afincado en Canadá, Geoffrey E. Hinton (76 años).
John J. Hopfield (91 años) desarrolló, en la década de 1980, redes neuronales sencillas que generaban, a partir de un conjunto de datos, paisajes de energía capaces de almacenar “recuerdos” en sus valles. Estas redes permitían reconocer patrones presentes en piezas de información incompletas o defectuosas, que podían ser mejoradas por comparación con los recuerdos almacenados. Hopfield fue un físico biológico
excepcional que propuso en la década de 1970 modelos para el transporte de electrones entre biomoléculas y para la corrección de errores en las reacciones bioquímicas.
Geoffrey E. Hinton (76 años) desarrolló redes neuronales más complejas y procedimientos de entrenamiento que permiten el aprendizaje automático y son la base de las aplicaciones de IA con las que interactuamos de forma habitual: esas que reconocen nuestro rostro, nos proponen series en las plataformas audiovisuales, actúan de asistentes virtuales personalizados, nos ayudan a generar música o texto, analizan con precisión nuestras imágenes médicas, deciden qué noticias nos llegan al móvil (uf), o nos atienden (ejem) cuando llamamos para hacer una reclamación. Hace un año, Hinton abandonó su empleo en Google tras alertar de
Figura 1
Niklas Elmehed © Nobel Prize Outreach.
los graves peligros que plantea el acelerado y poco regulado avance de la inteligencia artificial y proponer que se pongan en marcha investigaciones urgentes sobre su seguridad.
Una de las aplicaciones recientes de las redes neuronales con mayor impacto en Biología es la predicción de la estructura tridimensional de una proteína a partir de su secuencia de aminoácidos. Desde que Anfinsen demostró alrededor de 1960 que las proteínas se pueden plegar fuera de la célula en un medio acuoso sencillo, averiguar la estructura tridimensional que adoptará una secuencia de aminoácidos
disuelta en agua ha constituido uno de los grandes retos de la biología estructural. Como las proteínas son polímeros muy flexibles, identificar la conformación nativa funcional entre el elevadísimo número de conformaciones alternativas resulta muy difícil. Si la proteína es muy pequeña, es posible simular computacionalmente su plegamiento, pero, para la gran mayoría de las proteínas, la aproximación más exitosa ha sido, hasta hace poco, el modelado de su estructura por homología. Para ello se aprovecha que, si dos secuencias son parecidas, sus estructuras tridimensionales lo son todavía más. Mediante
el modelado por homología se puede predecir la estructura de una proteína si se ha determinado previamente la estructura de otra que tiene una secuencia parecida. Sin embargo, a pesar de que se han resuelto experimentalmente más de 200.000 proteínas, hay todavía muchas secuencias para las que no se conoce una estructura adecuada en la que basar su modelización. No obstante, las estructuras ya resueltas sí han resultado suficientes para entrenar una red neuronal artificial que consigue generar estructuras tridimensionales correctas. Esta red neuronal conocida como Alphafold (Figura 2) ha derrotado a todas las

Figura 2
Figura 3 del artículo: Jumper, J. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021). https://doi.org/10.1038/s41586-021-03819-2.
A FONDO
aproximaciones basadas en modelado por homología que laboriosa y atinadamente nos han permitido avanzar durante décadas, y que han sido rápidamente sustituidas por Alphafold. De repente, disponemos de modelos de bastante buena calidad de todas las proteínas humanas y de muchas otras especies, lo que facilitará, entre otras cosas de interés, la invención de nuevos fármacos o la modelización integral de la célula. Alphafold ha sido diseñada por la empresa DeepMind, adquirida por Google en 2013. Medio Premio Nobel de Química de este año ha sido concedido “por la predicción de la estructura de las proteínas” a Demis Hassabis, cofundador y CEO de Google DeepMind, y a John Jumper, director de la misma.
John Michael Jumper (39 años) es un científico estadounidense con sólida formación en Física y en Química que leyó su Tesis doctoral en 2017. Su título: New methods using rigorous machine learning for coarse-grained protein folding and dynamics ya daba que pensar. Demis Hassabis (48 años) comenzó diseñando videojuegos con un ZX Spectrum 48 comprado a los 14 años con sus ganancias como ajedrecista. Disfrutó de una breve y exitosa carrera científica como neurocientífico y fundó DeepMind. Hassabis también está preocupado por la regulación de la IA y, como Hinton, declaró en 2023 que “Mitigar el riesgo de extinción por IA debería ser una prioridad mundial junto a otros riesgos a escala social como las pandemias y la guerra nuclear”. Está claro que los mayores expertos en IA se toman en serio la posibilidad de que una Inteligencia Artificial General (un sistema tan o más capaz que una persona en la mayoría o en todas las actividades intelectuales) tome el control. ¿Alguien recuerda 2001: Una odisea del espacio?
Predecir la estructura de las proteínas a partir de su secuencia es grandioso, pero no deja sin trabajo a los biólogos estructurales que también desean poder crear proteínas nuevas que lleven a cabo nuevas tareas o las hagan mejor que las proteínas naturales. La importancia de las proteínas en biotecnología, biomedicina y en química sostenible es extraordinaria. Si la IA gestiona datos, las proteínas gestionan materia. Aprender a obtener proteínas a la carta sería una revolución con aplicaciones espectaculares. Este deseo creativo se plasmó en el desarrollo de la ingeniería de proteínas en la década de 1980, mediante la aplicación de técnicas de la biología molecular y con un ojo puesto en la comprensión de la termodinámica de las proteínas. Con la ingeniería de proteínas hemos aprendido a perfeccionar, seleccionar e incluso diseñar proteínas completamente nuevas. En el esfuerzo realizado, se percibe la tensión habitual entre el desarrollo de técnicas poderosas que no generan mucho conocimiento, pero sí productos de gran interés, y el esfuerzo paralelo, a menudo más lento, por llegar al mismo resultado generando, de paso, conocimiento científico. El acceso a nuevas proteínas se puede conseguir por dos vías extremas: generando muchas secuencias y seleccionando las que resulten adecuadas o mediante la aplicación de modelos físicos rigurosos que calculen de forma inversa qué secuencia va a dar lugar a una estructura con las propiedades que se desean. La primera aproximación está ilustrada por las técnicas de evolución dirigida que fueron galardonadas con el Nobel de Química en 2018. La segunda aproximación recibe este año la otra mitad del Nobel de Química 2024 “por el diseño computacional de proteínas” y celebra los logros del científico estadounidense David Baker (62
años) a quien la Wikipedia define como bioquímico y biólogo computacional. David Baker ha desarrollado su carrera científica en torno a los grandes problemas de la biología estructural: entender el plegamiento de las proteínas, predecir su estructura tridimensional y desarrollar métodos computacionales para el diseño de nuevas proteínas. Se ha caracterizado por llevar a cabo vigorosamente tanto estudios computacionales como experimentales (con frecuencia coordinados). En 2003, su grupo diseñó, utilizando métodos computacionales que habían desarrollado previamente, p. ej. Rosetta, la proteína Top7 que, según demostraron experimentalmente, adoptaba, de acuerdo a su diseño, un nuevo plegamiento de tipo α/ß desconocido hasta entonces. La proteína tenía una secuencia nueva, no inspirada por secuencias conocidas previamente, y se comportaba en disolución como era esperable. A partir de ese hito, su grupo ha trabajado en el diseño de nuevas proteínas con funciones valiosas, tales como antígenos para la obtención de vacunas, pequeñas proteínas antivirales, proteínas para inmunoterapia o enzimas glutenlíticas. Algunos de estos diseños han dado lugar a empresas posteriormente adquiridas por grandes farmacéuticas. Los métodos computacionales desarrollados por Baker han liderado y avanzado singularmente el campo del diseño de proteínas. Aunque estos métodos no permiten todavía el diseño automático de proteínas nuevas o con nuevas funciones, han demostrado que hay una ruta muy prometedora, basada en el conocimiento físico de las proteínas, que conduce a su diseño exitoso, y auguran un futuro en el que inventaremos las proteínas necesarias para sanarnos y gestionar la Tierra apretando el botón de INTRO.


¿PODEMOS MEJORAR NUESTROS PÓSTERES? ¡SÍ, PODEMOS!
Carlos Gancedo Ex-Profesor de Investigación del CSIC
Las presentaciones en pósteres han recorrido un largo camino desde sus primeros pasos en la Biochemical Society en 1967, su internacionalización en la VI reunión de FEBS en Madrid en 1969 y su entrada en Norteamérica en el Biochemistry/Biophysics Meeting en 1974. Lo que la revista Science tituló en esa fecha “Poster Sessions: a new look at scientific meetings”, se ha convertido en una forma de presentación usual muy apreciada de comunicación científica.
Visitar una sesión de pósteres puede ser una estimulante experiencia o generar una cierta desilusión. La realidad suele ser una mezcla de ambas sensaciones, con mayor o menor inclinación hacia una u otra según los casos. La desilusión se genera al ver la cantidad de buen trabajo científico presentado de una forma que no llega a una parte importante de la asistencia, y eso es grave, para la asistencia, y para el crédito de los presentadores.
Sobre las causas de esa disfunción, se ha escrito, y se sigue escribiendo, como muestran los resultados de una búsqueda en Internet poniendo “How to prepare a scientific poster”. Entre ellos menciono, como muy útil e ilustrativo, el que se encuentra en este enlace: https://plasticity.webs. upv.es/ewExternalFiles/Rules%20 posters.pdf
A pesar de ello, a menudo, la presentación es tal, que parece dirigida al círculo de personas conocedoras del asunto o relacionadas con el presentador. Presentadores jóvenes, poco o mal orientados, no consideran que una sesión de pósteres ofrece la oportunidad de discutir sus resultados, no solo con conocidos, sino con personas ajenas al tema, y que puede servir para establecer inesperadas colaboraciones. La sesión de pósteres se desarrolla en medio de un conjunto de personas que se mueve, genera ruido, se distrae, y
cuya atención hay que captar compitiendo con otros. El visitante que no esté interesado de antemano en el póster no suele dedicar más de dos minutos a considerarle; hay que ofrecer algo que capture su interés.
Uno de los factores que hacen que un póster se ignore es la sobrecarga de material; a veces parece que más que informar al público se trata de impresionarle con la cantidad de trabajo realizado. Se olvida el principio KISS, keep it short and simple; menos es, a menudo, más. Como el espacio es limitado, hay que ser selectivo en el material presentado. Hay que transmitir un mensaje corto y claro que pueda ser captado fácilmente y “llevado a casa”. El presentar demasiado material impide captar el mensaje, hace que el texto se escriba en una letra pequeña con escasa visibilidad a poca distancia, lo cual no atrae la atención. Se aconseja que el tamaño de letra permita la lectura a una distancia de 1-1,5 m.
El póster debe ser comprensible en ausencia del presentador; el objetivo del tiempo de exposición previa es que un visitante se entere del contenido para que después, en la sesión de presentación, pueda discutir el material con el presentador. El título es muy importante; Alberto Sols decía que el título de un artículo científico “debe ser conciso, descriptivo, y completo en lo posible”; esto es igualmente aplicable al de un póster. Pensemos en un título corto, presentado con un tamaño de letra grande. Sigue una breve introducción al problema considerado y una sucinta descripción del abordaje usado en su estudio. En los resultados, presentar sólo los necesarios para que se capte y comprenda el mensaje que se quiere transmitir. Usar subtítulos para las distintas secciones y canalizar el flujo de información para facilitar su seguimiento, incluso utilizando flechas si fuese necesario.
Las ilustraciones, deben servir para ilustrar, no para llenar espacio. Largas listas de genes o proteínas solo suelen hacer lo último, y suelen hacer renunciar a continuar la visita. El color del fondo es a veces un problema. Se aconseja usar preferentemente fondo blanco, y se desaconsejan fondos de colores chillones u oscuros. Algo importante que no se suele considerar; es el espacio en blanco, o espacio negativo. Se trata de un espacio entre los distintos apartados que ayuda a su comprensión y evaluación independiente. En algunas páginas de Internet se sugiere que hasta un 30% de la superficie debería ser espacio en blanco. La decisión sobre cuánto, es difícil, pero es importante considerarla. Una conclusión final recogiendo el mensaje central cierra bien un póster informativo.
La preparación de un póster lleva tiempo y pensamiento lógico, y debe hacerse colocándose mentalmente en la posición del público visitante.
Peter N. Campbell, uno de los impulsores de FEBS que fue también Presidente del Comité de Educación de IUBMB, publicó hace casi cincuenta años, en la revista Biochemical Education - actualmente Biochemistry and Molecular Biology Education - un artículo en cuyo título se leía “A plea for better slides”. Creo que nos beneficiaríamos todos si aplicásemos una súplica similar a los pósteres y prestásemos atención a una serie de líneas maestras no muy difíciles de seguir. Un póster atractivo no convierte en buena una mala investigación; pero uno mal presentado dificulta reconocer una buena.
Agradezco a la Dra. CarmenLisset Flores (IIBM Sols-Morreale, Madrid), al Prof. Miguel A. Blázquez (IBMCP, Valencia) y a la Dra. Juana M. Gancedo (Ex-profesora de investigación, CSIC) la lectura crítica de una versión inicial de este texto.
RESUMEN Y CONSIDERACIONES
FINALES EXTRAÍDAS DEL SIMPOSIO DE EDUCACIÓN EN EL MARCO DEL 46º
CONGRESO DE LA SEBBM
Marina Lasa
Universidad Autónoma de Madrid. Coordinadora del Grupo de Educación de la SEBBM
El Simposio de Educación asociado al 46o Congreso de la SEBBM tuvo lugar en A Coruña el 3 de septiembre de 2024, siendo un evento al que concurrieron, no sólo docentes en el ámbito universitario de la enseñanza de la bioquímica y la biología molecular, sino también estudiantes de máster y doctorado de diversas disciplinas relacionadas con las biociencias moleculares.
La sesión dio comienzo con la bienvenida a los asistentes y la presentación de las ponencias seleccionadas por parte de la coordinadora del Grupo de Educación, que hizo un especial hincapié en la importancia de la reflexión sobre planteamientos docentes novedosos que permitan impulsar y afianzar un proceso educativo centrado en el estudiantado como pieza esencial de su aprendizaje.
“Transformando la enseñanza en bioquímica y biología molecular: un viaje a través de metodologías docentes innovadoras”
La primera intervención corrió a cargo del Dr. Manuel Becerra Fernández, de la Universidade da Coruña y perteneciente al grupo docente EXPRELA, que examinó el impacto de diversas metodologías educativas innovadoras implementadas en colaboración con otros docentes, con el fin de mejorar la calidad del aprendizaje de los estudiantes de diferentes asignaturas de grado y posgrado.
En primer lugar, el ponente nos habló del uso estratégico de screencasts, que son vídeos cortos acerca de temas propuestos por los profesores, en los que se graba parte o la totalidad de la pantalla del ordenador, acompañados por audios explicativos. Esta actividad se desarrolla en la asignatura optativa “Fundamentos Bioquímicos de Biotecnología”, de cuarto curso del Grado en Biología, individualmente o en parejas, que deben exponer en un tiempo limitado ante el resto de sus compañeros el tema escogido mediante el apoyo audiovisual generado con el programa de captura de vídeo Screencast-o-matic1, utilizando el formato

PechaKucha2, tras lo que se abre un turno corto de preguntas. De las respuestas a una encuesta que se realizó a través de Moodle, se deduce que, aunque el formato utilizado parece tener ciertas limitaciones, esta actividad permite que los estudiantes revisen conceptos complejos a su propio ritmo, reforzando su comprensión mediante ejemplos visuales.
A continuación, el Dr. Becerra reflexionó acerca de los beneficios de la utilización de rúbricas electrónicas, generadas con la herramienta de libre acceso Corubric3, para realizar una evaluación más objetiva y colaborativa entre los docentes y los estudiantes en diversas asignaturas del Grado en Biología y del Máster en Biología Molecular, Celular y Genética. Una tercera aproximación docente que se presentó en esta ponencia, y que resultó de gran interés para la audiencia, consiste en una actividad de AprendizajeServicio implementada en la asignatura de “Trabajo de Fin de Grado” del Grado en Biología, que
se basa en la elaboración por parte de los estudiantes de video-presentaciones cortas o infografías sobre temas que cumplen con los objetivos de desarrollo sostenible (ODS) de promoción de la salud y, por tanto, resultan de interés para la sociedad. Una vez elaborados, dichos materiales son divulgados por las entidades colaboradoras conveniadas, AFACO4 o ASFEDRO5, y también en redes sociales. Aunque esta aproximación didáctica necesita un grado de planificación y de coordinación muy elevados, se ha detectado, como ventaja de su implementación, una mejora de la comprensión y de la retención de conceptos esenciales por parte de los estudiantes, así como un fomento de la concienciación social y del desarrollo de habilidades prácticas en este contexto.
Por último, el ponente mostró el uso de la analítica del aprendizaje en la asignatura optativa “Fundamentos Bioquímicos de Biotecnología” del Grado en Biología, a través de la puesta a
disposición en Moodle de una serie de recursos que permiten monitorizar el progreso de los estudiantes, identificando sus preferencias y áreas de dificultad. Entre los recursos empleados se encuentran presentaciones y vídeos de grabación asíncrona de los temas, así como material complementario, como recursos bibliográficos, enlaces a páginas web de interés, juegos de crucigramas o diferentes tipos de cuestionarios creados con el software Hot Potatoes6. Del análisis realizado a partir del “informe de actividades” del Moodle de la asignatura, en el que se puede consultar la información de cada estudiante en cuanto a recursos consultados, así como horas, frecuencias y franjas horarias de acceso, se pone de manifiesto que los estudiantes muestran preferencias por las presentaciones de los temas, seguidas de las actividades complementarias realizadas con Hot Potatoes y los vídeos de las clases, mientras que su interés por los otros recursos
EDUCACIÓN UNIVERSITARIA
es casi nulo. Todo ello ha permitido ajustar estrategias de enseñanza en tiempo real y optimizar los resultados educativos.
“El aprendizaje basado en investigación: una herramienta eficaz para impulsar la competencia compromiso social en estudiantes de biociencias”
La segunda ponencia corrió a cargo de la Dra. Lidia Ruth Montes Burgos, de la Universidad del País Vasco, que expuso el proyecto de innovación educativa denominado “Bi3oGela”, que está llevando a cabo con otros docentes de su departamento y que tiene por objeto la familiarización de sus estudiantes en el área de los ODS, mencionados anteriormente, a través de la sinergia entre las asignaturas “Tecnología del DNA Recombinante” y “Técnicas Instrumentales” de los Grados en Bioquímica y Biología Molecular y Biotecnología, respectivamente. La ponente comenzó haciendo un breve planteamiento del problema que habían detectado en las asignaturas de estudio durante los últimos cursos académicos y que se puede resumir en que sus estudiantes muestran un nivel de indagación muy bajo, lo que está directamente relacionado con su grado de autonomía. La actividad que nos presentó se lleva a cabo mediante una metodología de aprendizaje basado en investigación combinada con aprendizaje por indagación, que se comienza con el planteamiento de una pregunta que aborde un reto de la sociedad relacionado con uno o varios ODS y se completa con el diseño de una estrategia adecuada para dar respuesta a dicha pregunta. En concreto, los estudiantes, distribuidos en grupos, diseñan un proyecto de investigación que les permita sobreexpresar y purificar una proteína cuyo uso responda a un reto social, mientras que los
profesores asumen la labor de asistir al estudiantado en todas las etapas del proceso.
Un aspecto interesante de esta aproximación metodológica es que, dado que los estudiantes aprenden investigando, su aprendizaje atraviesa las mismas etapas que cualquier proceso de investigación, en el que el diseño experimental es crucial para alcanzar el objetivo. Así, la actividad comienza con la organización de los equipos y la distribución de los roles de cada miembro, para continuar con la elaboración de un cronograma de las actividades a realizar, que es supervisado por los profesores. En una segunda etapa, los grupos seleccionan la proteína con la que quieren trabajar y el reto a abordar. Seguidamente comienza la fase de investigación, en la que se selecciona y se procesa la información relevante, se plantea el desarrollo metodológico y se alcanzan y se discuten los resultados. Finalmente, en la etapa de presentación del trabajo, cada grupo entrega un informe detallado, que se incluye en un libro de resúmenes y se presenta en forma de comunicación oral.
Por otra parte, además de recibir la formación estrictamente curricular relacionada con las asignaturas en las que se ha implementado esta actividad, en paralelo y a lo largo de su desarrollo, se ofrecen talleres a los estudiantes sobre diversos aspectos que les pueden resultar de utilidad, como, por ejemplo, búsqueda bibliográfica y gestión de referencias. Asimismo, se les ofrece un taller sobre la Agenda 2030 y los ODS, por parte de una experta en sostenibilidad de la Asociación del País Vasco para la UNESCO7, con el fin de que los estudiantes mejoren sus conocimientos en retos sociales.
En la última parte de su ponencia, la Dra. Montes nos mostró algunos de los indicadores de
los resultados obtenidos de la implementación de este abordaje, poniendo de manifiesto que los estudiantes aumentan sus conocimientos específicos de las asignaturas implicadas y demostrándose que el éxito no está asociado específicamente al estudio de contenidos, pues las pruebas de evaluación se realizan sin previo aviso. Además, se ha experimentado una mayor diversificación de los ODS de elección en los estudiantes que asisten al taller relacionado con los mismos. Por último, se ha visto que la actividad mejora la percepción de los estudiantes sobre sus conocimientos sobre la Agenda 2030 y los ODS, constituyendo, por tanto, una herramienta eficaz para trabajar la competencia transversal relacionada con el compromiso social en biociencias y contribuyendo, de ese modo, a la formación de egresados que sean capaces de integrar desafíos sociales, ambientales y económicos desde un punto de vista del desarrollo sostenible.
Debate y conclusiones del simposio
En el debate que se estableció al terminar cada una de las ponencias, los asistentes al simposio formularon preguntas acerca de diversos aspectos relacionados con las mismas. Por ejemplo, en referencia al uso de screencasts, se discutió sobre la complejidad de los temas en los que se basan en relación con la formación de los estudiantes a los que van dirigidos, llegándose a la conclusión de que la elección del tema debe depender del perfil, del curso y del grado de madurez de los estudiantes. Además, se hizo mención sobre la importancia de la monitorización del trabajo individual en actividades que se realizan en parejas o grupos, para que el sistema de evaluación aplicado sea lo más justo posible. También se estableció un debate interesante al tratar acerca de los métodos
de evaluación y retroalimentación que se aplican con el fin de fomentar el aprendizaje. En este sentido, el ponente indicó que este punto constituye un área de mejora del uso de esta herramienta con fines docentes, ya que no ha sido posible proporcionar una retroalimentación muy completa debido al elevado número de estudiantes que participan en esta actividad.
Por otra parte, en cuanto a la ponencia de la Dra. Montes, el diálogo se centró en temas como el tipo y la formación previa de los estudiantes a los que van dirigidas este tipo de aproximaciones basadas en el aprendizaje por indagación, así como en la amplitud de la pregunta a abordar, llegándose al consenso de que ésta debe ser lo suficientemente precisa para evitar la divagación innecesaria por parte de los estudiantes. Además, también se plantearon otras cuestiones relacionadas con el sistema de evaluación aplicado en función de la participación de los estudiantes en los talleres complementarios sobre la agenda 2030 y los ODS. En este sentido, se hizo hincapié en que, independientemente de que los estudiantes hubieran recibido o no esa formación, la actividad descrita mejora su percepción sobre sus conocimientos a este respecto y, además, es valorada muy positivamente porque la consideran como una aproximación similar a situaciones a las que se pueden enfrentar en su futuro profesional. Por último, el simposio finalizó con un resumen de las principales conclusiones por parte de la coordinadora, que hizo un especial énfasis en la importancia de la realización de este tipo de reuniones en las que los docentes compartan sus experiencias educativas, todo ello con la finalidad de optimizar el aprendizaje activo de nuestros estudiantes y su preparación para el mercado laboral.
Otras comunicaciones
El Simposio de Educación se completó con la presentación de una serie de comunicaciones en forma de póster, en las que se describen diversas estrategias del proceso de enseñanza y aprendizaje. Por una parte, se mostraron dos propuestas de creación de vídeos interactivos aplicados a la enseñanza de las asignaturas “Bioquímica” y “Prácticas integradas de biología molecular y celular”, en formato de aula invertida, en los que hay diversas preguntas incrustadas que deben ser contestadas por los estudiantes para continuar la visualización. En ambos casos, la contestación de dichas preguntas contabiliza un 10% en la evaluación final de las asignaturas y, además, en la segunda de ellas, los estudiantes deben impartir un seminario de grupo, que contabiliza un 5% de la calificación final. Ambas aproximaciones muestran una valoración positiva por parte de los estudiantes, observándose una mejora del éxito académico en relación con el de cursos en los que se emplearon metodologías más tradicionales. Por otro lado, también se presentó una actividad de aprendizaje basado en proyectos diseñada para estudiantes del Grado en Biotecnología y de un curso de Farmacología, con
Referencias
https://screencast-o-matic.com/
la finalidad de evaluar la eficacia de un fármaco desde diferentes puntos de vista, en la que los estudiantes se involucraron muy activamente, ya que se les había informado previamente de que su resultado estaría vinculado e integrado en los contenidos del resto de los cursos. Por último, es de resaltar la presentación de una actividad de participación pública incluida en la Feria de Ciencias organizada por la Sociedad de Investigadores Españoles en el Reino Unido (SRUK), en el marco de su simposio anual en Glasgow (2018), cuyo principal objetivo fue explicar el estilo de vida de un parásito dentro del contexto del sistema inmunológico, facilitando su comprensión a través de la humanización de la explicación al comparar la célula con una casa y estableciendo una analogía entre los orgánulos de la célula y los componentes de la casa, para explicar sus funciones. Esta aproximación, que se completó con una actividad lúdica consistente en que los asistentes pudieran pintar su propio parásito mediante el uso de sellos preformados de orgánulos y crear su pieza artística mediante el uso de técnicas de impresión, tuvo una gran acogida por el público asistente.
https://www.adayapress.com/wp-content/uploads/2018/04/ched20.pdf
https://corubric.com/
https://afaco.es/alzheimer-investigacion-universidad-es.html
https://asfedro.org/es/infodrogas
https://hotpot.uvic.ca/
https://unetxea.org/
INVESTIGACIÓN

Un gradiente de sodio transmitocondrial controla el potencial de membrana en mitocondrias de mamífero
Pablo Hernansanz-Agustín, Carmen Morales-Vidal, et al. 2024. A transmitochondrial sodium gradient controls membrane potential in mammalian mitochondria. Cell S0092-8674(24)00974-7. doi: 10.1016/j.cell.2024.08.045
La función y la supervivencia de las células eucariotas dependen del uso de un gradiente mitocondrial electroquímico de protones, que se compone de un potencial de membrana mitocondrial interna y un gradiente de pH. Dicho gradiente electroquímico es generado por la función de la cadena de transporte de electrones, que transfiere los electrones de los sustratos provenientes de la dieta al oxígeno. Este proceso ocurre de forma acoplada a un bombeo de protones de un lado a otro de la membrana. Hasta ahora, se suponía que el potencial de membrana mitocondrial estaba compuesto exclusivamente de protones. Un estudio publicado en Cell, realizado por el grupo GENOXPHOS (CNIC y CIBERFES) en colaboración con los grupos de Proteómica Cardiovascular (CNIC y CIVERCV) y el Laboratorio de Membranas Mitocondriales (Universidad Complutense), ha descubierto que el potencial de membrana mitocondrial también está compuesto por un gradiente de sodio. Utilizando una colección de modelos genéticos mitocondriales y nucleares, demuestran que el gradiente de sodio es paralelo al de protones y puede llegar a constituir hasta la mitad del potencial de membrana mitocondrial en mamíferos. El gradiente de sodio se forma por la actividad del intercambiador sodio/protón, cuya identidad molecular se ha buscado desde hace casi seis décadas y lo habían identificado como el módulo P del complejo I. La desregulación de esta función, sin afectar la actividad enzimática canónica o el ensamblaje del CI, ocurre en la neuropatía óptica hereditaria de Leber (LHON), lo que desemboca en una profunda afectación del potencial de membrana mitocondrial y la homeostasis del calcio celular explicando así la patogénesis molecular previamente desconocida de esta enfermedad neurodegenerativa.

Papel esencial del receptor de folato en
el
rejuvenecimiento de neuronas del hipocampo
Alejandro Antón-Fernández, Rocío Peinado Cauchola, Félix Hernández, Jesús Ávila. 2024. Hippocampal rejuvenation by a single intracerebral injection of one-carbon metabolites in C57BL6 old wild-type mice. Aging Cell e14365. doi: 10.1111/acel.14365
En 2006, Yamanaka fue galardonado con el Premio Nobel por demostrar que no solo es posible que una célula embrionaria se diferencie en otros tipos celulares, sino que el proceso inverso también puede ocurrir en cultivos celulares, mediante la expresión de los factores que llevan su nombre. Inspirados en estos trabajos, el grupo liderado por J. Ávila (CBM ‘Severo Ochoa’, CSIC/UAM), en un artículo publicado en Aging Cell, ha utilizado una mezcla de cuatro metabolitos (putrescina, glicina, treonina y metionina) que inyectaron en el giro dentado de ratones viejos. El giro dentado es una región cerebral esencial para la función de la memoria y es extremadamente vulnerable al envejecimiento. Los resultados indican que en el tratamiento de ratones viejos con dicha mezcla se volvían a expresar receptores como GluN2B, que son específicos de ratones jóvenes. Además, ponen de relieve que el mecanismo de acción de estos metabolitos se basaría en la interacción de los mismos con el receptor alfa del folato (que se expresa en neuronas y apenas en otros tipos celulares); tras su internalización, induciría la transcripción de los factores de Yamanaka, por tanto, promoviendo procesos de rejuvenecimiento neuronal y funcional, puesto de manifiesto por un incremento de la capacidad cognitiva de los ratones viejos. En resumen, para inducir el rejuvenecimiento neuronal pueden utilizarse compuestos simples que actúen como ligandos del receptor alfa del folato y, a su vez, los resultados permiten explorar estrategias terapéuticas para enfermedades cerebrales relacionadas con la edad.

El hipocampo envejecido revela un rejuvenecimiento en respuesta al enriquecimiento ambiental
Raúl F Pérez, Patricia Tezanos, et al. 2024. A multiomic atlas of the aging hippocampus reveals molecular changes in response to environmental enrichment. Nature Commun. 15(1):5829. doi: 10.1038/ s41467-024-49608-z
Nuestro estilo de vida puede tener un gran impacto en nuestro bienestar general, así como en el desarrollo de enfermedades a lo largo de la vida. Durante el envejecimiento, nuestro genoma acumula alteraciones epigenéticas que involucran a la metilación del ADN y modificaciones postraduccionales de histonas. Estas alteraciones, que no afectan directamente a la secuencia del ADN, sí pueden alterar la expresión de genes y desencadenar programas moleculares anómalos. El equipo de Mario F. Fraga y Agustín F. Fernández (CINN, CSIC) ha publicado un estudio en Nature Communications en el que han analizado múltiples capas moleculares del hipocampo de ratones jóvenes y viejos, logrando describir el proceso de alteración del epigenoma durante el envejecimiento y generando un atlas molecular del hipocampo dorsal murino que abarca, entre otras, expresión génica, niveles de proteínas, metilación del ADN, accesibilidad de la cromatina y modificaciones de histonas, que han permitido detectar múltiples cambios en la heterocromatina. Adicionalmente, se analiza el efecto de una intervención de enriquecimiento ambiental. En este sistema, los animales conviven durante meses con juguetes, rampas o túneles, lo que conlleva una estimulación física, cognitiva y social. Curiosamente, pudieron observar que muchos de los cambios asociados al envejecimiento revertían durante el enriquecimiento ambiental, particularmente involucrando a los oligodendrocitos. De esta manera, en este estudio se han detallado con una profundidad sin precedentes los cambios epigenómicos que acontecen durante el envejecimiento y también los mecanismos moleculares que explican cómo una intervención de estilo de vida puede atenuar las alteraciones moleculares que acumulamos a lo largo de nuestras vidas.

El tamaño celular determina un transcriptoma óptimo para el crecimiento
Pedro J Vidal, Alexis P Pérez, et al. 2024. Transcriptomic balance and optimal growth are determined by cell size. Molecular Cell 84(17):3288-3301.e3. doi: 10.1016/j.molcel.2024.07.005
Aunque se asume que las células ajustan el tamaño para optimizar su crecimiento y ejecutar programas de desarrollo específicos, las evidencias acumuladas hasta ahora son muy débiles. Por otra parte, las células alcanzan un tamaño crítico antes de iniciar transiciones clave del desarrollo o del ciclo celular, pero se desconocían las razones por las que un tamaño determinado representa una ventaja. El trabajo publicado en Molecular Cell aborda esta cuestión fundamental y observan que la tasa de crecimiento disminuye tanto en células de tamaño menor o mayor que el de células normales. A su vez, el transcriptoma se altera profundamente y sufre una inversión relativa en respuesta al tamaño celular. Mientras que la transcripción de genes más expresados disminuye con la masa celular, la de los genes menos expresados aumenta. Usando diversas aproximaciones tanto teóricas como experimentales, concluyen que el desequilibrio transcriptómico se debe a los cambios en RNA polimerasa (RNApol) relativos al genoma que ocurren al disminuir o incrementar la masa celular a ploidía constante. Así, una disminución o aumento de la disponibilidad de RNApol altera mucho más la expresión de genes con promotores poco afines que la de genes muy activos, ya de por sí muy saturados por RNApol. Finalmente, alteraciones transcriptómicas que mimetizan los efectos del tamaño celular producen una fuerte disminución de la velocidad de crecimiento. Estos datos permiten proponer que las células fijan un tamaño crítico para obtener un transcriptoma adecuadamente equilibrado y, como resultado, maximizar el crecimiento durante la proliferación celular. Al mismo tiempo, el trabajo aporta conclusiones valiosas para comprender los efectos nocivos de la hipertrofia celular en patología humana.
INVESTIGACIÓN

PocketVec:
bioinformática aplicada al descubrimiento de fármacos
Arnau Comajuncosa-Creus, Guillem Jorba, Xavier Barril, Patrick Aloy. 2024. Comprehensive detection and characterization of human druggable pockets through binding site descriptors. Nature Communications 15(1):7917. doi: 10.1038/s41467-024-52146-3
La identificación de sitios farmacológicamente accionables ha sido un desafío durante largo tiempo en la investigación biomédica. Tradicionalmente, la caracterización de estos sitios ha dependido de técnicas experimentales costosas y lentas, como la cristalografía de rayos X o la resonancia magnética nuclear. Recientemente, un estudio realizado por el equipo de Patrick Aloy en el IRB Barcelona ha presentado una herramienta computacional (PocketVec) que facilita la caracterización de "bolsillos farmacológicamente aprovechables", definidos como zonas hidrofóbicas en las proteínas donde pequeñas moléculas pueden unirse y modular su función. El estudio ha sido publicado en la revista Nature Communications y representa un avance significativo en la bioinformática aplicada al descubrimiento de fármacos. PocketVec emplea un enfoque innovador basado en el cribado virtual inverso de moléculas pequeñas, para generar descriptores numéricos de los sitios de unión de proteínas, facilitando comparaciones y análisis a gran escala. Estos descriptores permiten identificar similitudes entre bolsillos que no se pueden detectar mediante comparaciones basadas únicamente en la secuencia o la estructura de las proteínas. Además, al utilizar tanto estructuras proteicas experimentales como modelos generados por AlphaFold2, los investigadores han sido capaces de identificar y caracterizar más de 32.000 bolsillos, la mayoría en proteínas que previamente no habían podido ser analizadas. La base de datos generada por PocketVec constituye un recurso sin precedentes para la comunidad científica, ofreciendo una visión que va más allá de la simple secuencia genética para centrarse en las interacciones moleculares que pueden ser aprovechadas para el desarrollo de nuevos fármacos.

El complejo I mitocondrial de la microglía es esencial para el desarrollo correcto del
cerebro
Bella Mora-Romero, Nicolas Capelo-Carrasco, et al. 2024. Microglia mitochondrial complex I deficiency during development induces glial dysfunction and early lethality. Nature Metabolism 6(8):1479-1491. https://doi.org/10.1038/s42255-024-01081-0
El cerebro es un órgano fundamental que determina quiénes somos y nuestra capacidad de interpretar e interaccionar con el mundo exterior. El desarrollo de este órgano es un proceso altamente complejo donde intervienen distintos tipos celulares, entre ellos el sistema inmune innato cerebral, la microglía. Estas células actúan como “barrenderas” para eliminar las neuronas y las conexiones no funcionales, esculpiendo de manera fina y en función de la experiencia postnatal la funcionalidad del órgano. Los autores han abordado el estudio de la contribución de la mitocondria a la actividad de la microglía. En su artículo publicado en Nature Metabolism, describen que la pérdida del complejo I de la cadena de transporte de electrones no limita inicialmente la actividad fisiológica de la microglía, sino que incluso la estimula. Con el tiempo, estas células terminan siendo disfuncionales, produciendo finalmente la alteración de otras células cerebrales, deterioro cognitivo y la muerte temprana de los animales. Mutaciones similares se asocian en humanos con el síndrome de Leigh, una enfermedad primaria mitocondrial que afecta a 1 de cada 40.000 nacidos a nivel mundial. Este síndrome progresa con problemas neurológicos y estudios previos habían definido la relevancia de la microglía en la progresión de la enfermedad. Este trabajo va un paso más allá y señala a la microglía como una contribuidora directa a la enfermedad, abre nuevas dianas terapéuticas y define las ventanas de actuación en enfermedades primarias mitocondriales. Adicionalmente, los resultados pueden tener consecuencias en como interpretamos la neuro-inflamación subyacente a los procesos de neurodegeneración, y como la actividad de la microglía podría ser controlada en dichos procesos.
Kuhner TOM para el análisis de gases en matraces agitados
Schulte A.1,3, Maschke R.2, Laidlaw D.1, Anderlei T.1
1 Kühner AG, Birsfelden, CH 2 ZHAW, Wädenswil, CH
3 Autor correspondiente
IntroducciónIntroducción
Para facilitar y acelerar el desarrollo de procesos y medios, las técnicas de medición online para matraces agitados acompañan al muestreo manual para obtener la máxima información del cultivo. El análisis de los gases producidos por el cultivo proporciona la tasa de transferencia de oxígeno (OTR), la tasa de transferencia de dióxido de carbono (CTR) y el cociente respiratorio (RQ) como medidas cuantitativas del estado fisiológico del cultivo. A escala de matraz agitado, suelen realizarse múltiples cultivos en paralelo.
Por consiguiente, el análisis de los gases debe ser rentable, fácil de manejar y versátil para adaptarse a diversas aplicaciones. Por lo tanto, hemos desarrollado un sistema de análisis de gases del cultivo para la determinación en línea no invasiva de OTR, CTR y RQ. El TOM (Transfer rate Online Measurement) está construido de forma modular para el análisis en 4, 8, 12 o 16 matraces individuales. El nuevo TOM de Kuhner puede aplicarse a varios tamaños y tipos de matraces (con baffels, de plástico, de vidrio), lo que permite al usuario obtener información sobre sus procedimientos de cultivo actuales.


Reproducibilidad

S. cerevisiae, YEP-medium (20 g/L glucose), n = 250 rpm, Vflask = 250 mL, d0 = 25 mm, T = 30°C, diferentes volúmenes de solución (10%, 20%), se muestran los valores medios de 4 cultivos individuales con sus respectivas desviaciones estándar.
El crecimiento exponencial se produce durante las primeras 8 h a un RQ de 4-5, lo que indica producción de un producto reducido (por ejemplo, etanol). El consumo de glucosa lo indica la caída del CTR tras unas 10 h. Esto va acompañado de un cambiometabólico a un RQ de aprox. 0,65, lo que indica un crecimiento con un sustrato reducido (por ejemplo, etanol). Ambos cultivos sufren una limitación de oxígeno después de 22 h y 23,5 h. Esto permite calcular los valores de kLa para las respectivas condiciones de cultivo.
Comprensión del proceso Precisión

La precisión de las mediciones OTR y CTR depende del volumen de cultivo y de la frecuencia de medición (aquí 3/h). Depende ligeramente del rango de concentración debido a la no linealidad de la salida del sensor de CO2 Desviaciones medias estándar en la medición del OTR, CTR y RQ de 12 frascos individuales, T = 30°C, n = 200 rpm, d0 = 25 mm, 4% volumen de cultivo. Determinadas con un sistema de modelo a RQ = 1.
Adolf Kühner AG
Dinkelbergstrasse 1
CH - 4127 Birsfelden, Switzerland

E. coli, Biener medium (5 g/L glycerol, 5 g/L ribose), n = 300 rpm, Vl = 4%, Vflask = 500 mL, T = 30°C, d0 = 25 mm. Valores medios de 4 cultivos individuales con sus correspondientes desviaciones estándar.
E. coli se cultivó en medio Biener definido que contenía 5 g/L de glicerol y 5 g/L de ribosa para investigar la fuente de C preferida y las respectivas tasas de crecimiento. Las condiciones de agitación no limitaban el oxígeno, ya que no se aprecia saturación en OTR. Sorprendentemente, no se observa crecimiento diauxico, lo que indica que tanto el glicerol como la ribosa se metabolizan simultáneamente. Esto se complementa con el RQ medido, que no cambia significativamente a lo largo del cultivo y coincide con el RQ teórico para el consumo simultáneo de ambas fuentes de C. El RQ se calcula como el cociente entre el CTR y el OTR medidos. La precisión es, por tanto, función de la precisión y el ruido de estas medidas. Va mejorando con una mayor respiración (véase el apartado «precisión de medición»).
Conclusión
El análisis de gases producidos por los cultivos a escala de matraz agitado, aporta diversa información que acelera el desarrollo del medio y el proceso y el escalado. De los datos extraídos del análisis de gases podemos obtener: La tasa de crecimiento, consumo y limitación de sustratos, el pH e inhibición de sustratos, formación e inhibición de productos, equilibrio de dióxido de carbono, limitación de oxígeno, kLa y condiciones de funcionamiento out of phase.
Kuhner TOM es un nuevo y versátil sistema de análisis de gases que permite al usuario recoger esta información con mediciones OTR, CTR y RQ de alta precisión.
Referencias:
A Aidelberg G Towbin B. D., Rothschild, D., Dekel E., Bren, A., & Alon, U. (2014). Hierarchy of nonglucose sugars in Escherichia coli https://doi.org/10.1186/s12918-014-0133-z
B Anderlei, T Zang W Papaspyrou M & Büchs J. (2004). Online respiration activity measurement (OTR, CTR, RQ) in shake flasks. https://doi.org/10.1016/S1369-703X(03)00181-5
C Von Stockar, U , & Liu, J. S. (1999). Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth. https://doi.org/10.1016/S0005-2728(99)00065-1
Reconocimiento

ANTONIO SILLERO (1938-2024)

Carlos Gancedo
Ex-Profesor de Investigación del CSIC
Nos ha dejado Antonio Sillero, excelente científico y docente, respetado y querido en los sitios por donde pasó. No me es fácil escribir esta nota sin sentir una importante pérdida personal; Antonio era mi amigo desde nuestra época de doctorandos en el grupo de Alberto Sols en el inolvidable CIB de Velázquez. Formábamos parte de “la generación de los sesenta” con Pepe y Marisa Salas, y Juana María Sempere (hoy Gancedo). Recorrí con él y su hermano menor, en 1965, un entonces casi intransitado Camino de Santiago y eso nos unió más todavía. Proveniente de Granada, donde estudió Medicina y Ciencias Químicas, Antonio se incorporó - tras una breve estancia, por motivos logísticos, en el laboratorio de Gabriela Morreale y Francisco Escobar- al grupo de Enzimología de Sols en 1964. Inicialmente trabajó en la regulación por insulina de la síntesis de la glucokinasa y otras enzimas de la glucolisis y la gluconeogénesis; trabajos con los que obtuvo los doctorados en las materias de sus estudios universitarios; el de Ciencias Químicas en Madrid y el de Medicina en Granada.
Durante ese periodo se incorporó a esos trabajos -en principio temporalmente- María Antonia Günther, procedente del grupo de José L. Rodríguez-Candela. La temporalidad se transformó en algo duradero; casi desde ese momento María Antonia fue compañera de Antonio en su vida investigadora y familiar. Entre 1967 y 1970 Antonio y María Antonia trabajan en el laboratorio de Ochoa en temas diferentes. Antonio trabaja sobre el bacteriófago Qβ e inicia trabajos sobre Artemia salina, un crustáceo de fácil cultivo. Vuelven a España y desde esa fecha las referencias que haga a la labor investigadora de Antonio tienen a María Antonia como co-protagonista. Prosigue trabajos en el desarrollo de Artemia y sus estudios prolongados producen importantes resultados sobre el metabolismo del dinucleósido polifosfato Gp4G, la interconversión de purín nucleótidos y las enzimas implicadas en esas reacciones. Su gran curiosidad hace que trabaje en otros temas como metabolismo y función de bisfosfonatos, simulación matemática de procesos biológicos, regulación de encrucijadas metabólicas y evolución de
proteínas, realizando estancias, incluso en la década final de su actividad, en la Universidad de Barcelona y en el University College de Londres.
Antonio se interesó por la docencia universitaria, y mediante oposición obtuvo la plaza de agregado de Fisiología y Química biológica en la Universidad de Valladolid en 1975. Cuatro años después es catedrático de Bioquímica en la Universidad de Extremadura en Badajoz, donde permanecerá ocho años que consideraba “los más fructíferos y felices de su vida investigadora”; allí fue Vicerrector de Investigación y Director del Instituto de Ciencias de la Educación. En su etapa final vuelve como catedrático a la Facultad de Medicina de la UAM donde da su última clase
Antonio Sillero falleció el día 4 de octubre de 2024 después de una larga enfermedad que le ha tenido postrado casi cuatro años. El papel de María Antonia en el mantenimiento de Antonio durante estos años tan dolorosos ha sido heróico y fundamental. La labor conjunta de Antonio y María Antonia (A+A) ha cesado después de una vida matrimonial dedicada a la investigación científica. La conjunción de estas dos personalidades ha dado como fruto unos resultados que quedan reflejados en la excelencia y el gran número de artículos científicos publicados y sobre todo en la gran cantidad de discípulos que por ellos han sido formados en investigación bioquímica. Y más importante aún, ha sido la estela de humanidad que esta pareja ha dejado en todos los ambientes en que su vida se ha desarrollado. Han sido verdaderos hacedores de Paz
Antonio nace en Rute (Córdoba) y se licencia en Medicina en la Universidad de Granada y su afición por la bioquímica le lleva a licenciarse también en Ciencias Químicas en la misma Universidad que le permitirá conocer más profundamente los procesos bioquímicos. Finalizadas sus Licenciaturas pide la incorporación en el grupo de investigación que dirige el Dr. Alberto Sols en el Centro de Investigaciones Biológicas de la calle de Velázquez de Madrid. La falta de espacio en este Departamento de Enzimología, le obliga a esperar por un año su incorporación al mismo. Los Drs. MorréaleEscobar le ofrecen su laboratorio durante este periodo de espera. En el Departamento de Enzimología, y dirigido por el Dr. Sols, realiza el trabajo experimental para obtener sus doctorados en Medicina y Ciencias
en 2008. Sus clases dejaban huella en sus alumnos; como muestra cito el recuerdo, décadas después, de una alumna destacada, Cristina Lamas, quien evoca “sus explicaciones poniendo énfasis en la finalidad de las reacciones metabólicas para las funciones vitales, así como su fantasía para atraer a los alumnos”.
La ciudad de Rute (Córdoba), de la que era nativo, le otorgó en 2011 el premio “Villa de Rute” a la Cultura, Ciencia e Innovación, premio que, sin duda, le llegó muy hondo.
En el recuerdo de muchas personas que conocieron a Antonio, surge como característica cumbre, su gran humanidad, su disposición a oír y a ayudar. Así queremos recordarte, amigo Antonio.
Químicas. Antonio y María Antonia realizan trabajo postdoctoral en el departamento de Bioquímica que dirige el Profesor Severo Ochoa en la New York University. Vueltos a España, Antonio dirige sus pasos hacia la enseñanza universitaria opositando a la cátedra de Bioquímica de la Universidad de Valladolid, y ejerciendo después enseñanza en la Universidad de Extremadura en Badajoz. Su paso por ambas Universidades dejó poso abundante tanto en personal como en número y calidad del trabajo de investigación desarrollado, gracias al esfuerzo tanto suyo como la eficaz participación de María Antonia. Badajoz cuenta hoy con un competente Departamento de Bioquímica en la Facultad de Medicina gracias a la eficaz acción de esta pareja. Finalmente terminó su carrera como catedrático en la Facultad de Medicina de la Universidad Autónoma de Madrid.
Escribo estas líneas con el sentimiento de dolor que aún hoy, a los quince dias del fallecimiento de Antonio, me produce su pérdida, pero con el consuelo de haber compartido con él (léase A+A) tantos y tantos momentos de mi vida. Las inmemorables fiestas en su domicilio con participación de amigos y vecinos en la puesta a punto de conciertos musicales y representaciones teatrales son inolvidables. Su origen andaluz hacía que todos estos actos terminasen con el canto de la Salve Rociera que tan emotivamente sonó en el momento de su enterramiento. ”Morir sólo es morir. Morir se acaba. Morir es una hoguera fugitiva. Es cruzar una puerta a la deriva y encontrar lo que tanto de buscaba” (Martín Descalzo). Seguro que Antonio encontró ya esa Paz de la que fue ejemplar difusor.
Claudio Fernández de Heredia Socio ordinario jubilado de la SEBBM
PREMIOS DE INVESTIGACIÓN SEBBM 2024
PREMIO IBUB-SEBBM

Marina García Macía
El premio que concede la SEBBM en colaboración con el Instituto de Biomedicina de la Universidad de Barcelona (IBUB), en reconocimiento a la trayectoria investigadora de las socias y socios jóvenes de la SEBBM, ha recaído en su edición 2024 en la investigadora Marina García Macía del Instituto de Biología Funcional y Genómica (IBFG)/Universidad de Salamanca-CSIC. El premio está dotado con 2.500 €. La galardonada ofreció la conferencia Autophagy to rule them all el pasado jueves 5 de septiembre en el 46º Congreso de la SEBBM en A Coruña.
PREMIO ‘PROFESORA MARÍA TERESA MIRAS’ AL MEJOR ARTÍCULO DE JÓVENES DE LA SEBBM
El premio reconoce los mejores artículos científicos publicados por los jóvenes investigadoras/es SEBBM. En esta edición se han concedido un premio de 1.000 € y un accésit de 500 €.
María Jesús González Rellán
El primer premio ha sido para María Jesús González Rellán (CiMUS, Departamento de Fisiología, Universidad de Santiago de Compostela) por el artículo Neddylation of phosphoenolpyruvate carboxykinase 1 controls glucose metabolism publicado en la revista Cell Metabolism 35, 16301645.e5 (2023), doi: 10.1016/j.cmet.2023.07.003. La Dra. González Rellán, primer autor del artículo, impartió una conferencia sobre este trabajo el 5 de septiembre en el 46º Congreso de la SEBBM celebrado en A Coruña.
Ayelen Melina Santamans Recchini (Accésit)
El accésit ha sido para Ayelen Melina Santamans Recchini (Centro Nacional de Investigaciones Cardiovasculares (CNIC), Madrid) por el artículo MCJ: A mitochondrial target for cardiac intervention in pulmonary hypertension publicado en la revista Science Advances 10, eadk6524 (2024), doi:10.1126/sciadv.adk6524.
PREMIO JOSÉ TORMO



La SEBBM en colaboración con BRUKER Española ha concedido el premio, en la edición de este año, a Marina Serna (Centro Nacional de Investigaciones Oncológicas (CNIO), Madrid) por su trabajo Transition of human γ-tubulin ring complex into a closed conformation during microtubule nucleation publicado en la revista Science 383:870-876 (2024), doi: 10.1126/science.adk6160. El premio, dotado con 1.000 €, se concede al mejor artículo científico, publicado por un socio/a joven investigador/a, entre el 15 de junio de 2023 y el 15 de junio de 2024 en cualquiera de las disciplinas que engloba la Biología Estructural. Marina Serna impartió la charla Cryo-EM structure of the gamma-Tubulin Ring Complex reveals the mechanism for de novo microtubule en la sesión conjunta de los grupos Biomembranas - Estructura y función de proteínas el 5 de septiembre en el pasado 46º Congreso de la SEBBM celebrado en A Coruña. Desde SEBBM felicitamos a las socias premiadas.
ENTREGA DE PREMIOS FUNDACIÓN BBVA-SEBBM A JÓVENES
INVESTIGADORES 2024
El pasado 6 de septiembre se entregaron los premios Fundación BBVA - SEBBM a las mejores comunicaciones orales y/o póster presentados durante el 46º Congreso SEBBM por las y los socios adheridos (doctorandos o doctores recientes – dos años máximo desde la lectura de la tesis doctoral), en cada uno de los dieciséis grupos científicos temáticos de la sociedad. El Acto tuvo lugar durante la ceremonia de clausura del 46º Congreso SEBBM.
Los galardonados, en esta edición, han sido Laura Salmerón Pelado (Hospital Universitario de Tarragona Joan XXIII, Pere Virgili Health Research Institute (IISPV), Tarragona) del grupo “Bases moleculares de la patología”; Raquel Flores-Hernández (Instituto de Neurociencias de Castilla y León e Instituto de Investigación Biomédica de Salamanca) del grupo “Biología del desarrollo y Modificación genómica”; Almudena Saavedra Bouza (Centro Interdisciplinar de Química y Biología (CICA), Universidad de A Coruña) del grupo “Biología molecular ómica y Bioinformática”; Daniel Abella López (CiQUS-Centro
Singular de Investigación en Química, Biología y Materiales moleculares de Santiago de Compostela, A Coruña) del grupo “Biología sintética y Biotecnología molecular”; Juan Ortiz Mateu (Universidad de Valencia) del grupo “Biomembranas”; Andrea Alegre Martí (Instituto de Biomedicina, Universidad de Barcelona) del grupo “Estructura y Función de proteínas”; Amanda Guitián Caamaño (Grupo CELLCOM, Centro de Investigación Biomédica (CINBIO) e Instituto de Investigación Biomédica de Orense (IBI), Pontevedra-Vigo) del grupo “Mitocondria, Comunicación celular y Estrés oxidativo”; Elena Lavado Fernández (Instituto de Biología y Genómica (IBFG), Universidad de Salamanca/CSIC) del grupo “Muerte celular e Inflamación”; Diego González Pérez (Centro de Investigación en Medicina Molecular y Enfermedades Crónicas (CiMUS) de Santiago de Compostela, A Coruña) del grupo “Neurobiología molecular”; Oihane Altube (Departamento de Química Aplicada de la Universidad del País Vasco (UPV/EHU), Donostia, Guipúzcoa) del grupo “Parasitología
molecular e Infecciones emergentes”; Ana Rodríguez Fernández (Grupo de Terapia celular e Ingeniería de tejidos (TERCIT), Instituto de Ciencias de la Salud, Universidad de Ias Islas Baleares) del grupo “Química biológica”; Marcos Jiménez Juliana (Centro de Biología Molecular (CBM) ‘Severo Ochoa’, Madrid) del grupo “Regulación de la expresión génica y Dinámica del genoma”; Anna Marsal-Beltrán (Hospital Universitario de Tarragona Joan XXIII, Pere Virgili Health Research Institute (IISPV), Tarragona) del grupo “Regulación metabólica y Nutrición”; Naroa GoikoetxeaUsandizaga (CICbioGUNE, Derio, Vizcaya) del grupo “Senescencia celular”; y Antonio García
Estrada (Instituto Maimónides de Investigación Biomédica de Córdoba (IMIBIC), Córdoba) del grupo “Señalización celular”. El premio correspondiente al grupo “Metabolismo del nitrógeno y Bioquímica de plantas y microorganismos” ha quedado desierto en la edición 2024.
Desde SEBBM felicitamos a las premiadas y premiados.

PREMIO CERTEST BIOTEC A LA MEJOR IMAGEN CIENTÍFICA DEL AÑO 2024

ENTREGA DE MEDALLAS A LOS SOCIOS DE HONOR
SEBBM 2023
El pasado 6 de septiembre se entregaron las medallas a los “Socios de Honor” de la SEBBM nominados en 2023. Las Prof. Dras. Caroline Dean (John Innes Centre, Reino Unido) y Mª Ángeles Serrano (Universidad de Salamanca) recibieron este reconocimiento por la labor que cada una de ellas ha desarrollado para la promoción de la investigación, el fomento de la transferencia del conocimiento, el impulso a los jóvenes científicos, la expansión internacional de la ciencia, y la difusión y divulgación en la sociedad. La distinción fue propuesta por la Comisión de Admisiones de la SEBBM, presidida por el profesor Félix M. Goñi Urcelay, y aprobada por la Asamblea General de socios celebrada durante el 45º Congreso SEBBM, en septiembre de 2023, en Zaragoza.
El premio CerTest Biotec a la mejor Imágen científica del año 2024 se falló durante el pasado 46º Congreso de la SEBBM con las votaciones de las socias y socios. Al concurso concurrieron las 12 imágenes seleccionadas y publicadas en la “Pinacoteca de la Ciencia” de la SEBBM entre julio de 2023 y junio de 2024. La ganadora, con mayor número de votos, fue la imagen titulada "La espada del rey Arturo, edición cristalográfica” de Aurora Martín González (Instituto de Biomedicina y Biotecnología de Cantabria (IBBTEC), Santander), publicada en marzo de 2024. La imagen representa la forma ordenada en la que se organizan las proteínas, formando estructuras cristalinas. La autora escribe: “en este caso los cristales nos muestran formas tan curiosas como las de la fotografía, una prueba más de que en la ciencia también puede haber arte”. La entrega del premio tuvo lugar durante la ceremonia de clausura del 46º Congreso SEBBM.

Mª Ángeles Serrano recibió la medalla en la sesión de clausura del 46º Congreso de la SEBBM en A Coruña. La Prof. Dean no pudo estar presente en la ceremonia. Recibió la medalla unos días antes en su domicilio y manifestó su enorme agradecimiento. La Comisión de Admisiones, en la convocatoria de 2024, ha propuesto “Socios de Honor” a la Dra. Isabel Fabregat Romero (IDIBELL, Barcelona) y al Dr. Roger Davis (EEUU). Ambos reconocimientos fueron aprobados en la Asamblea General de socios celebrada durante el 46º Congreso SEBBM en A Coruña.

ISABEL FARIÑAS PREMIO NACIONAL DE INVESTIGACIÓN 2024
La Dra. Isabel Fariñas, socia SEBBM, ha sido distinguida con el Premio Nacional de Investigación ‘Santiago Ramón y Cajal’ 2024, en el ámbito de la Biología, que concede el Ministerio de Ciencia, Innovación y Universidades. El premio reconoce sus originales aportaciones a la biología de los factores neurotróficos y las células madre neurales, y sus aplicaciones al tratamiento de enfermedades neurológicas que han contribuido al progreso y bienestar de la sociedad. Sus aportaciones científicas son reconocidas tanto en el ámbito nacional como internacional. Sus trabajos pioneros en el área de la neurogénesis adulta han obtenido un proyecto del Consejo Europeo de Investigación ERC Advanced Grant 2022. Isabel Fariñas es catedrática de Biología Celular, e investigadora principal y coordinadora del grupo de Neurobiología Molecular de la Universitat de València desde 1998. Su grupo de investigación pertenece al CIBER en Enfermedades Neurodegenerativas (CIBERNED) y es grupo de excelencia Prometeo de la Comunidad Valenciana. Ha sido miembro de la junta directiva de las sociedades españolas de Neurociencia (SENC), de Terapia Génica y Celular y de Biología del Desarrollo (SETGYC), así como de la International Society of Differentiation Desde 2013 es miembro de la prestigiosa European Molecular Biology Organization (EMBO) y en 2014 fue elegida por la Fundación Botín-Banco Santander para formar parte de su programa de ciencia para la incentivación de la transferencia tecnológica. Es vocal del patronato de la Fundación Carmen y Severo Ochoa (FCySO).


46º CONGRESO DE LA SEBBM EN A CORUÑA
María D. Mayán Santos
Presidenta
del 46º Congreso SEBBM
CINBIO.
Universidad de Vigo
El 46º Congreso de la SEBBM se celebró en A Coruña durante la primera semana de septiembre, y ha sido un evento memorable, que reunió a cerca de 700 inscritos y 30 empresas participantes. Este congreso nacional, de carácter internacional, permitió disfrutar de investigaciones de vanguardia realizadas tanto en España como en el extranjero. La conferencia inaugural fue impartida por Tak Mak, quien compartió sus últimos avances en inmunoterapias para el cáncer y su potencial aplicación en enfermedades neurodegenerativas. El congreso culminó con una conferencia de Evan Rosen, quien presentó resultados recientes sobre la biología del tejido adiposo, abriendo nuevas perspectivas y funciones. A lo largo del congreso, los simposios abordaron una amplia gama de temas, desde la quiescencia hasta la aplicación de inteligencia artificial en la biología molecular, mostrando avances prometedores que ya están teniendo impacto en investigación. Este año, los grupos científicos unieron esfuerzos para optimizar recursos, lo que permitió reducir el número de salas y los costes del congreso. La SEBBM es un congreso extenso, con múltiples sesiones paralelas, lo cual representa un desafío logístico y a menudo dificulta la asistencia a todas las charlas de interés. Sin embargo, la alta calidad de los ponentes y de las presentaciones asegura una experiencia enriquecedora tanto a nivel académico como personal. En el 46º congreso contamos con un equipo de voluntarios formado por predoctorales y postdoctorales de grupos SEBBM, quienes colaboraron para garantizar el

buen funcionamiento de todas las actividades. Además, se implementó un espacio de cuidado infantil, patrocinado por la Xunta de Galicia, facilitando la conciliación familiar y permitiendo a las y los asistentes participar plenamente en el congreso. Asimismo, se llevaron a cabo actividades de divulgación científica, con gran éxito, en colaboración con entidades públicas y privadas, como la Fundación Carmen y Severo Ochoa, la Asociación Española contra el Cáncer A Coruña, la Fundación San Rafael, la GAIN y la ACIS entre otras. Estas actividades, dentro de lo que conocemos como “Bioquímica en la ciudad”, contribuyen a la transferencia de conocimiento, a volorar la ciencia por parte de la sociedad y dan una mayor proyección al congreso. Entre las actividades satélites, la sesión del Consejo Europeo de Investigación (ERC), en la que María Leptin y varios beneficiarios de proyectos ERC compartieron sus experiencias y consejos, animó a presentarnos a estas convocatorias, tanto a los más jóvenes como a los más senior. También me gustaría destacar las sesiones en las que entidades privadas, como la Fundación FERO y la Fundación Pasqual Maragall, entre otras, explicaron sus planes de financiación
y captación de talento, reflejando el interés y el auge de la inversión privada en ciencia e innovación en España, que ha crecido en estos últimos diez años.
La encuesta de satisfacción realizada una vez finalizado el congreso a los participantes proporcionó comentarios valiosos que ayudarán a mejorar la organización de ediciones futuras. Aunque el desafío de las sesiones paralelas dentro del propio congreso es complejo de resolver. No me gustaría terminar este artículo sin mencionar el curso de iniciación a la investigación, la sesion de innovación y la sesión de mujer y ciencia, sesiones muy bien valoradas e importantes dentro de cada congreso SEBBM. Organizar un congreso de esta magnitud requiere un esfuerzo continuo durante un año, con una intensidad especial en los últimos meses. Sin embargo, la experiencia y los resultados finales hacen que todo ese esfuerzo valga la pena. Con gran ilusión, esperamos el 47º Congreso de la SEBBM, que se celebrará en Cáceres, organizado por Guadalupe Sabio. Tendrá lugar la primera semana de septiembre del próximo año, en un entorno histórico en el centro de la ciudad. Espero veros allí para agradeceros personalmente y daros un cariñoso abrazo.
HISTORIA VIVA DE LA BIOQUÍMICA ESPAÑOLA EN DIRECTO:
El profesor José Antonio Cabezas nos escribe carta
Una parte muy importante de la bioquímica española actual puede trazar su genealogía a unos pocos laboratorios de la postguerra civil española. Entre ellos, quizá el más temprano con reconocimiento onomástico fue el de Ángel Santos Ruiz (https://dbe.rah.es/biografias/21757/angel-santos-ruiz), pues D. Ángel ostentó desde 1940, en la Facultad de Farmacia de la Universidad entonces llamada Central y hoy Complutense de Madrid, la primera cátedra con la denominación específica de Química Biológica. Esta denominación de la bioquímica era entonces común, reflejándose aún hoy en la de la revista Journal of Biological Chemistry (nacida en 1905). Dado el origen tan temprano del laboratorio de D. Ángel y su centralidad (recuérdese que por entonces la Universidad Central era una de las pocas que expedía títulos de doctor), no es de extrañar que dicha cátedra fuera un importante nodo filogenético de la bioquímica española.
De dicho nodo original procede José Antonio Cabezas, a quien me referiré hoy aquí brevemente por haber recibido una carta suya en la que dice ser (seguramente

con razón) el ex-catedrático vivo de Bioquímica más antiguo de nuestro país. No me extenderé en narrar su prolongada peripecia personal (95 años de plena lucidez), pues su biografía se resume en la web de la Real Academia de Farmacia (https://ranf.com/academico/cabezas-fernandez-del-campo-j-antonio/), Academia de la que D. José Antonio es Académico numerario; y también, en más detalle, en la Wikipedia (https://es.wikipedia.org/wiki/ Jos%C3%A9_Antonio_Cabezas_ Fern%C3%A1ndez_del_Campo). Sí rememoraré a instancias suyas que, al frente de su grupo en la Universidad de Salamanca, realizó en los años 70 y 80 contribuciones cruciales a la caracterización de numerosas glicosidasas. Bastantes de ellas forman parte de la clasificación de la Enzyme Commission gracias a su trabajo de descripción, identificación, caracterización y distinción con respecto a otras. Entonces la discriminación basada en secuencia completas aún no era ni siquiera una posibilidad, y la basada en genética se restringía a algunos microorganismos y levaduras. Me envía el Dr. Cabezas unas cuantas referencias ilustrativas,
que adjunto como imágenes. Cuando se es muy longevo es muy raro no ser testigo de la desaparición de algún hijo científico. Este ha sido para el Prof. Cabezas el caso de su discípulo Prof. Ángel Reglero, catedrático que fue de Bioquímica y Biología Molecular de la Universidad de León y Organizador del XXV Congreso SEBBM, celebrado en esa ciudad en 2002, siendo uno de los Congresos SEBBM de más concurrencia y éxito que recuerdo (incluido el XXXVIII Congreso, que organicé yo mismo en Valencia en 2015). Dice también el Dr. Cabezas en su carta que esta nota debería ser un homenaje póstumo a Ángel Reglero, coautor clave en algunos de esos ya viejos trabajos. Comparto esa idea. Ya antes hube de compartir con el Dr. Cabezas y con su nieta científica, Prof. María Ángeles Serrano, el obituario por Ángel Reglero en esta revista (SEBBM Nº 206; Diciembre 2020, pg. 42-43), así que, también yo, brindo estas líneas a mi fallecido amigo.
Vicente Rubio Zamora Miembro del Comité editorial de la Revista SEBBM

Figura 1
José Antonio Cabezas (Cortesía de la Real Academia de Farmacia)
Figura 2
Fragmento de la carta autógrafa del Prof. Cabezas
PREMIOS NOBEL EN FISIOLOGÍA O MEDICINA 2024
El Premio Nobel de Fisiología o Medicina 2024 ha sido para los genetistas estadounidenses
Victor Ambros y Gary Ruvkun por el descubrimiento de una clase de pequeñas moléculas, los micro-ARN, que tienen un papel crucial en el control de la expresión genética.
“Su revolucionario descubrimiento reveló un principio completamente nuevo de regulación génica que resultó ser esencial para los organismos pluricelulares, incluido los humanos. Ahora se sabe que el genoma humano codifica más de mil micro-ARN”, destaca la Academia sueca.
Victor Ambros, trabaja en la Facultad de Medicina de la Universidad de Massachusetts en Worcester, y Gary Ruvkun en el Hospital General de Massachusetts (MGH) en Boston.
Regulación de la actividad de los genes
Comprender la regulación de la actividad de los genes ha sido un objetivo importante durante mucho tiempo. En la década de 1960 se demostró que proteínas especializadas, conocidas como factores de transcripción, pueden unirse a regiones específicas del ADN y controlar el flujo de información genética determinando qué ARNm se produce. Desde entonces, se han identificado miles de factores de transcripción, y durante mucho tiempo se creyó que se habían resuelto los principios fundamentales de la regulación génica. No obstante, en 1993, los galardonados con el Nobel de este año publicaron hallazgos inesperados que describían un nuevo nivel de regulación génica, que resultó ser muy significativo y conservado a lo largo de la evolución.
Ambros y Ruvkun, publicaron sus primeros descubrimientos clave en 1993 cuando trabajaban como investigadores posdoctorales del mismo grupo. Identificaron dos genes que intervienen en el desarrollo del gusano Caenorhabditis elegans. Las mutaciones en estos genes impedían que los embriones del gusano se desarrollaran adecuadamente. Trabajando en laboratorios separados, Ambros se sorprendió al descubrir que uno de los genes no codificaba una proteína, sino que producía una cadena de ARN curiosamente corta. El trabajo de Ruvkun sobre el segundo gen que sí codificaba una proteína, ayudó a completar el cuadro. Los investigadores descubrieron que la cadena del ARN, posteriormente llamada micro-ARN, se unía al ARN mensajero codificante de la proteína, impidiendo su traducción.
Durante años, el descubrimiento se consideró una peculiaridad exclusiva de los gusanos, sin mucha relevancia para otros organismos.
Sin embargo, esta visión cambió en 2000, cuando el equipo de Ruvkun identificó otro micro-ARN de C. elegans que, era compartido por humanos, ratones y la mayor parte del resto del reino animal. El descubrimiento de que los micro-ARN se conservan en todo el árbol de la vida ha llevado a una intensa investigación en este campo.
Estudios en ratones con genes codificadores de micro-ARN mutados demuestran que estas moléculas tienen funciones cruciales en el desarrollo, la fisiología, y el comportamiento entre otras actividades.
Si bien las posibilidades terapéuticas de los micro-ARN son todavía lejanas, los investigadores esperan algún día aprovechar estos reguladores maestros para identificar y tratar enfermedades. https://www.nature.com/articles/ d41586-024-03212-9
El Premio Nobel de Medicina para Ambros y Ruvkun por el microARN | madrimasd

Figura 1
Victor Ambros (izquierda) y Gary Ruvkun (derecha)
2024: UN AÑO CON MUCHO RINCÓN DEL AULA
Como científicos tenemos el deber de transmitir nuestro trabajo a la sociedad. Hemos de explicar nuestros avances para que toda la población entienda el valor de mantener una investigación nutrida de inversión y grandes mentes. Desde la Sociedad Española de Bioquímica y Biología Molecular (SEBBM) realizamos diferentes actividades que fomentan este intercambio de conocimientos y que intentan nutrir a la población con ciencia contada en primera persona. Todo gracias a la voluntad incansable de sus protagonistas, los científicos. En el Rincón del Aula se abordan temas que necesitan ser trasladados a la población, temas cotidianos, como por ejemplo ¿qué es la senescencia? o ¿por qué es importante la microbiota? o más novedosos como la autosis. El Rincón es un espacio abierto a la divulgación que encontramos en la web de la SEBBM, y que recoge pequeños artículos divulgativos dirigidos a la población general. Esta sección consigue captar la atención, principalmente de profesores de secundaria de una amplia distribución geográfica, desde nuestro país hasta toda Hispanoamérica.
En 2024, el Rincón ha tratado temas variados, desde la relevancia de la compartimentalización celular, hasta sistemas para generar ratones modificados genéticamente, muy útiles para estudiar procesos fisiológicos. Se ha hecho hincapié en muchos temas relacionados con la salud, como el envejecimiento, analizando la relevancia de combatir la senescencia para envejecer de forma más saludable. O la microbiota, crucial para el mantenimiento de nuestra salud gastrointestinal. E incluso, se ha reseñado una nueva estrategia para el desarrollo de

Figura 1
Rincón del Aula 2024. Creado en BioRender. Garcia-Macia, M. (2024) https://BioRender.com/x21r570
fármacos contra el cáncer y las enfermedades neurodegenerativas denominada PROTACs. Por otra parte, se han discutido temas tan interesantes como la organización de la información genética en los virus. Estos consiguen condensar su genoma para que ocupe un espacio muy limitado, y aunque parezca trivial, esto es relevante para el diseño de vacunas y ciertas terapias antivirales. También, se ha relatado la última forma de muerte celular, la autosis. Descubierta muy recientemente, se produce en alguna situación patológica como puede ser la hipoxia, e implica un reciclaje celular excesivo que ocasiona un desenlace fatal. Los siete artículos publicados este año vienen acompañados con unas figuras muy ilustrativas que facilitan la interpretación de la información y generan un material didáctico excelente.
La variedad temática muestra la diversidad de expertos de la SEBBM. Revela cómo la
bioquímica es una rama de la ciencia que se integra perfectamente en multitud de los ámbitos de estudio y responde a muchas de las demandas sociales de conocimiento. En este último año destaca la paridad de l@s autor@s, jóvenes investigadores con una trayectoria muy interesante, que sin lugar a duda darán muchas alegrías a la investigación de nuestro país. Por lo tanto, el Rincón del Aula tiene una importante función divulgadora, y también sirve como plataforma para promocionar a nuestros investigadores. Estos podrán incluir el artículo dentro de su portfolio divulgador ya que incluye su propio DOI, y, además, el artículo incluye su biografía y fotografía. Esto fomenta las relaciones interpersonales entre investigadores de la SEBBM. Para formar parte del Rincón del Aula, escríbenos a sebbm@sebbm.es (Figura 1)
Marina García Macia
Vocal Junta Directiva SEBBM
HAY BIOLOGÍA (Y MUCHA)
MÁS ALLÁ DEL ADN
Víctor de Lorenzo Departamento de Biología de Sistemas Centro Nacional de Biotecnología (CNB), CSIC

How life works. A user's guide to the New Biology | Philip Ball Picador | Londres (2023) | 541 p.
El último libro de Philip Ball, escritor científico con muchos años de rodaje en Nature, presenta una visión renovada de los sistemas vivos en el marco de lo que denomina la Nueva Biología, orientada a entender la vida desde la perspectiva física de los sistemas complejos y los fenómenos de emergencia causal. Para ello, el autor desafía la visión convencional centrada tradicionalmente en los genes como la base de todos los procesos vitales. Esta nueva interpretación cuestiona la primacía del ADN a base de analizar la existencia de propiedades, estructuras dinámicas y procesos en los organismos que no se derivan únicamente de sus genomas y de hecho se independizan claramente de ellos.
El punto de partida e hilo conductor de todo el texto es la revisión de la idea de la célula y de los sistemas vivos como máquinas cuyo funcionamiento sigue la misma lógica relacional entre sus componentes que un artefacto mecánico o eléctrico. El éxito histórico de esta metáfora tiene un gran apoyo en la biología molecular tradicional y el modelo del operón de Monod, que reproduce fielmente a escala molecular los dispositivos de retroalimentación bien conocidos por los ingenieros. Pero ahora Ball reformula la cuestión preguntándose qué distingue entonces a un sistema vivo de otro que no lo es sin caer en el vitalismo anterior a Pasteur. En este contexto, el autor especula con lo que llama vitalismo molecular, es decir, las características específicas de los entes biológicos que los separan del mundo abiótico. Como alternativa, propone una comprensión más profunda de los sistemas vivos a través de conceptos y métodos tomados fundamentalmente de la termodinámica, la computación y la teoría de redes. Un tema central es el concepto de emergencia causal, o la pregunta sobre cómo fenómenos en una micro-escala molecular pueden dar lugar a comportamientos complejos en una macro-escala, tanto en escenarios biológicos existentes como en procesos evolutivos. Esta pregunta converge con el otro gran tema del libro: la noción de agencia. Ball la concibe como la capacidad de los sistemas vivos para tomar decisiones o realizar acciones en beneficio propio, sin depender completamente del control genético. Esta agencia es vista como una forma de propósito en los sistemas vivos, sin caer en un vitalismo metafísico. Según Ball, esta agencia permite a los seres vivos navegar y adaptarse a las restricciones físicas y termodinámicas de su entorno, utilizando la información para desarrollar comportamientos complejos y adaptativos. Para ilustrar esto, utiliza la metáfora del demonio de
Maxwell sobre cómo la información puede revertir las leyes de la termodinámica. De hecho, los sistemas vivos acumulan y gestionan información (no sólo en el ADN) para estructurar y coordinar sus funciones. Esta idea lleva al autor a proponer que la vida puede entenderse mejor como un proceso de captura y despliegue de información ejecutado materialmente y de forma autónoma por distintos actores moleculares y celulares. La biología es, por tanto, mucho más que el Dogma Central. Si hubiera que resumir el mensaje del libro en una sola frase, esa sería No todo está en los genes o No todo está en el ADN. En este sentido, Ball se aleja cautelosamente de Dawkins y su gen egoísta y se acerca, quizás sin percatarse de ello, a la visión de las células (no los genes) de Alfonso MartínezArias como protagonistas del desarrollo, así como a la idea de epistasis estructural de las bacterias elaborada por Fernando Baquero.
Tras leer el libro no puedo dejar de pensar en la máxima posmoderna de que no hay hechos, sólo hay interpretaciones, que tanto saca de quicio a biólogos como Lewis Wolpert. Pero lo cierto es que los sistemas biológicos y el fenómeno de la vida han pasado históricamente por distintas interpretaciones puntuadas por grandes cambios de paradigma. El vitalismo que dominó durante siglos las ciencias de la vida fue refutado primero por la síntesis química de la urea por Wöhler en 1828 y un siglo más tarde (y definitivamente) por el descubrimiento del ADN, la elucidación del flujo de expresión génica y los experimentos de química prebiótica de Stanley Miller. El Dogma Central y el gen/DNA egoísta han sido los marcos habituales para explicar mecanicistamente multitud de fenómenos biológicos. Al menos hasta ahora. Lo que tímidamente comenzó a denominarse epigenética, abrió la caja de Pandora a otras realidades y mecanismos no geno-céntricos que operan sobre los sistemas vivos. Y en la base de todos ellos, aparecen las nociones de emergencia causal y agencia como cualidades que no pueden deducirse meramente del ADN pero que necesitan estudiarse y entenderse.
En definitiva, Ball sostiene que la así llamada Nueva Biología representa un avance significativo en nuestra comprensión de las ciencias de la vida en la medida que las acerca a la física de los sistemas complejos. Sin embargo, para bien o para mal, la biología está aún lejos de ser una ciencia tan dura y coherente como otras disciplinas. Y a la espera de la aparición de futuros paradigmas, probablemente lo vivo seguirá desafiando nuestras interpretaciones y marcos conceptuales durante bastante tiempo.

Automatización y Uso de Robots en Laboratorios
Elisabeth Casado, Product Manager
Eduardo Valle, Technical Service Dep. Life Science, Aplitech Biolab
La automatización y el uso de robots en laboratorios están transformando significativamente la manera en que se realizan investigaciones científicas y pruebas de diagnóstico. Estos avances tecnológicos permiten realizar tareas complejas y repetitivas con mayor precisión, eficiencia y seguridad, mejorando así la productividad y reduciendo el riesgo de errores humanos. Sin embargo, la implementación de estas tecnologías también presenta desafíos que deben ser considerados.
BENEFICIOS
Precisión y Consistencia: Los robots pueden realizar tareas repetitivas con alta precisión y sin error humano, lo que reduce errores y aumenta la reproducibilidad de los experimentos.
Eficiencia y Productividad: Los robots pueden operar 24/7 sin necesidad de descansos, incrementando significativamente la productividad del laboratorio.
Seguridad: Los robots pueden manejar sustancias peligrosas y realizar tareas en entornos peligrosos, reduciendo el riesgo de exposición para los técnicos y científicos.
Costes Operativos: Aunque la inversión inicial puede ser alta, la automatización puede reducir gastos a largo plazo al disminuir la necesidad de personal y los errores.
Análisis de Datos: Los sistemas automatizados pueden integrarse con software avanzado para el análisis de datos, facilitando la recopilación, almacenamiento y análisis de grandes volúmenes de datos.
A TENER EN CUENTA
Inversión Inicial Alta: La adquisición e instalación de equipos automatizados y robots pueden ser costosos.
Flexibilidad Limitada: Los robots están diseñados para tareas específicas y han de desarrollarse nuevas aplicaciones para nuevos métodos o experimentos imprevistos.
Dependencia de la Tecnología: Los laboratorios pueden volverse altamente dependientes de la tecnología, y cualquier fallo en el sistema puede causar interrupciones significativas en el trabajo.
Requerimientos de Mantenimiento y Capacitación: Los equipos automatizados requieren mantenimiento regular y el personal necesita capacitación especializada para operar y mantener los sistemas.
La automatización y el uso de robots en laboratorios representan una poderosa herramienta para mejorar la precisión, eficiencia y seguridad de las operaciones. Siendo crucial equilibrar términos de costes, flexibilidad, y mantenimiento. La decisión de automatizar depende de las necesidades específicas del laboratorio, el tipo de investigaciones realizadas y los recursos disponibles.
Nuestros compañeros del Servicio Técnico de Aplitech nos aportan su opinión:
La automatización constituye una herramienta poderosa para evolucionar ya que elimina tareas repetitivas y rutinarias de las tareas diarias de los técnicos de laboratorio. Siempre que vamos a un cliente para realizar un mantenimiento de los diferentes equipos de automatización que representamos (Scinomix, Analytik Jena, Hamilton y Eprep) notamos, lo que llamamos, el efecto lavavajillas.
El efecto lavavajillas consiste en la experiencia familiar de las dificultades que tuvimos para que nuestras abuelas empezaran a utilizar el nuevo sistema de automatización de limpieza de utensilios de cocina, comúnmente conocido como lavavajillas. Para ellas, era un momento realizar la tarea y no suponía ningún esfuerzo tener que limpiar todos los cacharros sucios que se acumulaban después de cada comida, “esto no me hace falta” decían. Pero una vez cogida la costumbre se convirtió en un instrumento imprescindible que ahorraba tiempo y consumo de agua y energía, ya no entraba en la cabeza la idea de tener que realizar la tarea repetitiva y rutinaria que el protocolo decía que después de cada comida todo limpio de nuevo a mano.
Cuando visitamos a un cliente y preguntamos cómo está funcionando el equipo y si lo están utilizando, las respuestas son las mismas siempre, “el equipo va muy bien, nos ahorra mucho tiempo y ya ni nos imaginamos tener que hacer lo que hacíamos antes.”

Los equipos que representamos son equipos robustos con una larga vida útil, que pueden realizar largos ciclos de trabajo sin fallas y proporcionando unos resultados repetitivos y fiables durante muchas muestras. Para mi esa es la principal ventaja de este tipo de equipos que hace que su principal desventaja (coste inicial alto) se vea eliminada rápidamente.
Desde Aplitech podemos asesorar para ver cómo mejorar los procesos productivos de vuestros laboratorios o plantas e incluso realizar cálculos para comprobar en cuantos años la inversión del equipo resulta amortizada. Ofreciendo equipos de dispensación de líquidos automáticos o robots, para todo tipo de laboratorios, desde los más pequeños que quieren empezar con sistemas sencillos, a laboratorios con los sistemas más avanzados ya existentes. Para ello contamos con marcas como Hamilton Robotics con el microLab Prep o el CyBio Felix de Analytik Jena.
Apostar por la automatización es ganar calidad en el laboratorio.

Proquinor te, proveedor oficial de Revvity en España
Revvity y Proquinor te unen fuerzas para hacer te llegar la tecnología bioquímica más puntera a nivel de ensayos fluorescentes: El HTRF.
¿Qué es el HTRF( Homogeneous Time Resolved Fluorescence)?
El HTRF ™ es una tecnología que combina la tecnología FRET con la medición de fluorescencia. Se puede utilizar en ensayos celulares o bioquímicos, siendo de gran utilidad para el descubrimiento de fármacos, bioterapia, investigación básica en oncología, en SNC y en trastornos metabólicos e inmunitarios.
Ventajas
Fácil de realizar: sin necesidad de lavado
Ahorro de tiempo y mano de obra
Múltiples campos de aplicación
Analiza y detecta complejos pequeños y grandes

Señalización celular
Cuantificación de citoquinas
¿Quieres saber más?
Aplicaciones

Fuera ruido: Elimina la fluorescencia de fondo
Analiza y detecta interacciones de alta y baja afinidad

Investigación CGCR Cuantificación de biomarcadores



Interacción de proteínas Epigenética Ensayos de kinasas
Solicita más información acerca de esta nueva tecnología a través de nuestra página: www.proquinorte.com
