

UNIVERSIDAD NACIONAL DE MOQUEGUA
















ESCUELA PROFESIONAL DE INGENIERÍA AMBIENTAL

‘’Año de la unidad, la paz y el desarrollo” 2023 ILO -

1. INTRODUCIÓN
En el presente trabajo se presenta la simulación del gel de agarosa para cepas bacterianas encontradas en suelos y aguas contaminadas por derrame de hidrocarburos. Es por ello, que se usa la técnica de electroforesis en gel de agarosa para separar moléculas de ADN, ARN o proteínas en función de su tamaño, forma y carga eléctrica.
SnapGene es un software de clonación que ayuda a planear, simular y documentar manipulaciones de ADN de las cepas bacterianas Además, posee una interfaz intuitiva que permite visualizar las secuencias de ADN, anotación de secuencias, edición de secuencias, clonación, proteínas y métodos de clonación comunes. Por ende, es muy usado por científicos a nivel mundial por la eficiencia del tratamiento de secuenciación de ADN.
El gel de agarosa es la más utilizada para analizar y caracterizar ácidos nucleicos de distintas procedencias, estos geles se comportan como un tamiz molecular y permiten separar moléculas cargadas en función de su tamaño y forma
2. OBJETIVOS
2.1 Objetivo General
❖ Realizar la simulación de electroforesis en gel de agarosa utilizando el software
SnapGene con los datos proporcionados de las cepas bacterianas aisladas en suelos y aguas contaminados del artículo “Aislamiento de bacterias con potencial biorremediador y análisis de comunidades bacterianas de zona impactada por derrame de petróleo en Condorcanqui – Amazonas - Perú”.
2.2 Objetivo Específico
❖ Aplicar conocimientos básicos sobre el funcionamiento e importancia del gel de agarosa en cepas bacterianas.
❖ Usar datos proporcionados del NCBI
❖ Adquirir el método del manejo del software SnapGene para realizar la práctica.
3. MARCO TEÓRICO
3.1 Software SnapGene
SnapGene es la forma más fácil de planificar, visualizar y documentar sus procedimientos diarios de biología molecular. Personalice la visualización de sitios enzimáticos, características, cebadores, ORF, colores de ADN y más. Permite el manejo de datos para escanear grandes secuencias de ADN con miles de funciones anotadas.
Ayuda a realizar inserciones, eliminaciones, reemplazos y cambios de mayúsculas y minúsculas. Cuando se copia y pega una secuencia, las características se transfieren automáticamente. Encuentre características comunes en su secuencia de ADN utilizando la extensa base de datos de SnapGene
Proporciona ventanas elegantes y ricas en información para simular una variedad de métodos comunes de clonación y PCR. Registra automáticamente las operaciones para crear un historial gráfico y almacena las construcciones ancestrales en el archivo final.
SnapGene permite exportar una secuencia a formato GenBank o FASTA, para luego se convierta a mapa o imagen de gel a formatos estándar para usar con otro software.

3.2 Electroforesis
La electroforesis es una técnica de laboratorio que se usa para separar moléculas de ADN, ARN o proteínas en función de su tamaño y carga eléctrica. Se usa una corriente eléctrica para mover las moléculas a través de un gel o de otra matriz. Esta técnica se basa en el diferente movimiento de las moléculas cargadas eléctricamente a través de la superficie hidratada de un medio sólido al hacer pasar una corriente sobre el medio, las moléculas con una carga negativa, se mueven hacia el ánodo (+), mientras que las moléculas con carga positiva, se ven atraídas hacia el cátodo (-).
3.3 Gel de Agarosa
La agarosa es un polímero lineal de galactosa y 3,6-anhidrogalactosa. El gel se obtiene disolviendo la agarosa en un buffer de TAE o TBE y se funde usando un microondas, hasta obtener una solución homogénea y transparente. La disolución se vacía en un molde y se coloca un peine (el peine forma unos pozos donde se depositan las muestras). Se deja enfriar para polimerizar para formar una matriz porosa variando el tamaño del poro según la concentración de agarosa contenida en la disolución. Además, la migración de los fragmentos de ADN variará dependiendo del tamaño, conformación molecular (lineal, circular, superenrollado), concentración de la agarosa en el gel, voltaje, dirección del campo eléctrico, presencia de colorantes añadidos que se intercalan en el ADN
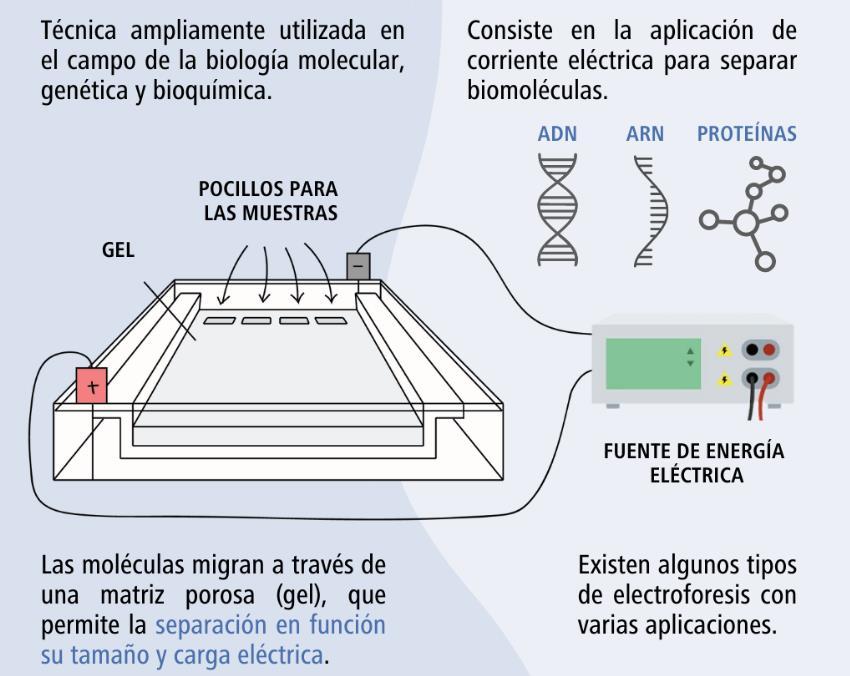
3.4 Electroforesis en Gel de Agorosa
La electroforesis en gel de agarosa es la técnica de separación más utilizada para el análisis y caracterización de ácidos nucleicos de distintas procedencias. Los geles se comportan como un tamiz molecular y permiten separar moléculas cargadas en función de su tamaño y forma. Así, moléculas de ADN de diferente tamaño y conformación van a migrar de forma distinta en una electroforesis en gel de agarosa.
La aplicación de marcadores de peso molecular (fragmentos de ADN de tamaño conocido) permite calcular el tamaño aproximado del ADN en estudio. En el caso de los geles de agarosa, se añade bromuro de etidio o algún colorante comercial (GelRed, GelGreen, SYBR Green…), substancias que se intercalan entre las bases del ADN y son fluorescentes cuando se ilumina con luz ultravioleta. Una vez finalizada la electroforesis se visualiza el gel con una lámpara de luz UV y se verán las bandas correspondientes a las muestras problema de ADN y los marcadores de peso molecular

3.5 Centro Nacional para la Investigación Biotecnológica (NCBI)

Es parte de la Biblioteca Nacional de Medicina de Estados Unidos, una rama de los Institutos Nacionales de Salud, fue fundado el 4 de noviembre de 1988 con la misión de ser una importante fuente de información de biología molecular. Almacena y constantemente actualiza la información referente a secuencias genómicas en GenBank, un índice de artículos científicos referentes a biomedicina, biotecnología, bioquímica, genética y genómica en PubMed, una recopilación de enfermedades genéticas humanas en OMIM, además de otros datos biotecnológicos de relevancia en diversas bases de datos. Todas las bases de datos del NCBI están disponibles en línea de manera gratuita, y son accesibles usando el buscador.
El NCBI que permite acceder a bases de datos de:
❖ Estructuras
❖ Secuencias de nucleótidos
❖ Secuencias de proteínas
❖ Genomas completos
4. METODOLOGÍA
4.1 Materiales
❖ Laptop
❖ NCBI
❖ Software SnapGene
❖ Artículo científico
❖ Carpeta de archivos (FASTA)
4.2 Método
En el presente trabajo de simulación de electroforesis mediante el gel de agarosa, se utiliza el software SnapGene y las cepas bacterianas del artículo científico.

PASOS:
1. Búsqueda de las cepas bacterianas en el NCBI, para ello copiamos el N de accesión de la cepa bacteriana y procedemos a buscar en el NCBI.


2. Descargamos la información del NCBI en formato FASTA

3. Descargamos todas las cepas bacterianas en formato FASTA

4. Abrimos los archivos descargados en el software SnapGene y colocamos en doble cadena y activamos la casilla

5. Después volvemos a abrir el software SnapGene para cargar el archivo descargado, para ello se debe presionar “OpenFiles” y aceptar.


6. Luego nos aparecerá la secuencia de ADN del primer dato cargado.

7. Ya al tener la secuencia, se procede a realizar la simulación del gel de agarosa, para ello se debe seleccionar en la barra de herramientas la opción “Tools” y presionar en ‘’Simulate agorose Gel’’.

8. Seleccionamos la simulación del gel de agarosa y nos aparece una nueva ventana donde se observa el rango PCR.

5. RESULTADO
El producto final de la simulación de gel de agarosa referente a los números de accesión del artículo científico de cepas bacterianas.
6. CONCLUSIÓN
❖ Se logró con eficiencia la práctica de simulación de electroforesis en gel de agarosa usando el programa de SnapGene.
❖ El software SanapGene, nos permite entender como se puede configurar las cepas bacterianas de acuerdo al porcentaje de agarosa y los elementos del gel.

7. BIBLIOGRAFÍA
❖ Electroforesis en gel. (s/f). Khan Academy.
https://es.khanacademy.org/science/ap-biology/gene-expression-andregulation/biotechnology/a/gel-electrophoresis
❖ ChecaRojas,A.(2017). Método: Gel deelectroforesis Agarosa.Conogasi, Conocimiento para la vida.
https://conogasi.org/articulos/metodo-gel-de-electroforesis-agarosa/
❖ Stryer L, Berg JM, Tymoczko JL (2003): “Bioquímica”, 5ª ed. Editorial Reverté (Barcelona, España)
❖ Archer SN, Schmidt C, Vandewalle G, Dijk DJ. Phenotyping of PER3 variants reveals widespread effects on circadianpreference,sleep regulation, and health.2018.
https://www.genome.gov/es/genetics-glossary/Electroforesis
❖ Centro_Nacional para la Información Biotecnológica (s/f). Quimica es
https://www.quimica.es/enciclopedia/Centro Nacional para la Infor maci%C3%B3n Biotecnol%C3%B3gica.html
❖ Sambrook J, Russel DW (2001). Molecular Cloning: A Laboratory Manual 3rd Ed. Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY.

