
6 minute read
BIOWISSENSCHAFTEN
from ChemieXtra 11/2020
by SIGWERB GmbH
Ribosomen sind die Proteinsynthese-Maschinen der Zellen. Sie lesen die genetischen Informationen von der Boten-RNA (roter Faden) ab und stellen darauf basierend Peptide (violette Fäden) her, die sich dann zu Proteinen falten.
«Deep Sequencing»
Advertisement
Wo die Ribosomen andocken
ETH-Forschende können für viele Milliarden verschiedene Abfolgen von RNA-Bausteinen vorhersagen, wie gut die zelluläre Proteinsynthese-Maschinerie an sie andockt. Dieses Andocken hat einen wesentlichen Einfluss darauf, wie viel von einem Protein hergestellt wird. Zur Entwicklung des Vorhersagemodells nutzen die Wissenschaftler eine Kombination von Experimenten der synthetischen Biologie und Algorithmen des maschinellen Lernens.
Fabio Bergamin ¹
Das Erbgut von Bakterien, Pflanzen bis hin zum Menschen lässt sich heute sehr einfach entziffern, und dennoch birgt es viele offene Fragen. Da sind zum Beispiel die RNA-Abschriften der Gene und die Stellen darauf, an welche die zelluläre Proteinsynthese-Maschinerie (die Ribosomen) andocken, um die genetische Information abzulesen. Ein umfassendes Verständnis zu diesen Andockstellen fehlte bisher. Ein interdisziplinäres Team von Forschenden des Departements Biosysteme (DBSSE) der ETH Zürich in Basel hat nun einen Ansatz entwickelt, mit dem es erstmals möglich ist, detaillierte Informationen zu einer unglaublich grossen Zahl solcher Andockungsstellen in Bakterien zu gewinnen. Der Ansatz kombiniert experimentelle Methoden der synthetischen Biologie mit maschinellem Lernen.
Feinsteuerung der Proteinherstellung
Bei den Andockungsstellen handelt es sich um kurze Abfolgen von RNA-Bausteinen vor jedem Gen. In der Vergangenheit haben Biotechnologen auch künstliche Andockstellen entwickelt. An einige davon heften sich die Ribosomen äusserst gut, an andere weniger. Je besser Ribosomen an eine bestimmte Variante andocken können, desto eher lesen sie die Information des Gens ab und desto mehr des entsprechenden Proteins stellen sie her. Wenn Biotechnologen Bakterien verwenden, um beispielsweise Medikamente herzustellen, können sie mit der Wahl der Ribosomen-Andockungsstellen die hergestellte Menge beeinflussen. «Besonders interessant und wichtig ist eine solche Steuerung, wenn man komplexe GenNetzwerke in Zellen bringt, die mehrere Proteine gleichzeitig herstellen sollen. Dann gilt es, ihre Menge optimal aufeinander abzustimmen», erklärt Markus Jeschek. Er ist Senior Scientist und Gruppenleiter am D-BSSE.
Ein Experiment mit 300000 Sequenzen
Zusammen mit den ETH-Professoren Yaakov Benenson und Karsten Borgwardt sowie Mitgliedern der jeweiligen Gruppen
Strukturaufklärung des Ribosoms Grundlegende Mechanismen während der Proteinbiosynthese (Translation) konnten erst dank einer umfassenden Strukturaufklärung dieser Zellmaschinerie verstanden werden. Dafür erhielten Venkatraman Ramakrishnan, Thomas A. Steitz und Ada E. Yonath 2009 den Nobelpreis für Chemie.

entwickelte er nun eine Methode, um für über 300 000 synthetisch erzeugte RNA-Sequenzen zu bestimmen, wie gut Ribosomen an sie andocken. Bisher war das pro Experiment nur für wenige Hundert Sequenzen möglich. Der Ansatz der ETH-Forschenden benutzt «Deep Sequencing», die modernste Technik zur Bestimmung von DNA- und RNA-Sequenzen. Im Labor stellten die Wissenschaftler und Wissenschaftlerinnen in einem Schritt mehr als 300 000 verschiedene Ribosomen-Andockungsstellen her und fusionierten diese jeweils mit einem Gen für ein Enzym, das ein Stück DNA verändert. Die entsprechenden Gen-Konstrukte führten sie in Bakterien ein, um im Experiment zu sehen, wie stark sich im Einzelfall die Ribosomen an die RNA heften. Je besser die jeweilige Andockungsstelle funktioniert, desto mehr Enzym wird in der Zelle hergestellt, und umso eher wird das Stück DNA verändert. Diese Veränderung können die Forschenden im Anschluss an das Experiment mittels Sequenzierung zusammen mit der jeweiligen RNA-Sequenz der Andockstelle auslesen.
Universeller Ansatz
Weil 300 000 nur ein kleiner Teil von vielen Milliarden theoretisch denkbaren Ribosomen-Bindungsstellen ist, haben die Wissenschaftler ihre Daten mit Algorithmen des maschinellen Lernens untersucht. «Diese Algorithmen erkennen in grossen Datenbeständen komplexe statistische Zusammenhänge. Mit deren Hilfe können wir vorhersagen, wie gut Ribosomen eine bestimmte RNA-Sequenz binden», erklärt Karsten Borgwardt, Professor für Data-Mining. Diese Vorhersagemodelle stellten die ETH-Forschenden als Software frei zur Verfügung, damit auch andere Wissenschaftler sie nutzen können. Bald werden sie auch einen einfach zu nutzenden Onlineservice veröffentlichen. Der von den Wissenschaftlern gewählte Ansatz ist universell, wie Benenson und Jeschek betonen. Deshalb planen die Forscher, ihn auch in anderen Organismen zu nutzen bis hin zu menschlichen Zellen. «Der Einfluss von genetischer Information auf die Menge eines Proteins, das in einer Zelle hergestellt wird, ist auch beim Menschen interessant», sagt Benenson. «Gerade auch im Zusammenhang mit genetisch bedingten Krankheiten.»
Originalpublikation S. Höllerer et al., «Large-scale DNA-based phenotypic recording and deep learning enable highly accurate sequence-function mapping», Nature Communications (2020); DOI: 10.1038/s41467-020-17222-4

Konzepte zu entwickeln, welche die Leistungsfähigkeit, Produktivität und Rentabilität Ihrer Anlage steigern, ist für Sie wichtig.
IDEENREICH + RISIKOARM
Wir unterstützen Sie verlässlich dabei, Produktqualität, Anlagensicherheit sowie Kosten- und Risikomanagement ganzheitlich zu betrachten.
Micropilot FWR30 – der cloudbasierte Füllstandssensor
• Transparent – umfassender und sicherer Zugriff auf
Bestandsinformationen, von überall und zu jeder Zeit • Einfach – von der Beschaffung bis zur Nutzung. Einfache
Inbetriebnahme, Bedienung und Optimierung von
Prozessabläufen • Flexibel – digitale Dienste für verschiedenste Anforderungen, von Netilion Value über Netilion
Inventory bis zu SupplyCare
Hosting • Zuverlässig – präzise Messung mit High-End 80 GHz-
Sensortechnologie,
Batteriebetrieb und mobile
Kommunikation
Erfahren Sie mehr unter: www.ch.endress.com/fwr30
Antibakterielle Cellulose
Ein Wundverband, der Bakterien abtötet
Damit bakterielle Infektionen direkt in der Wunde bekämpft werden können, haben Empa-Forschende Membranen aus Cellulose entwickelt, die mit antimikrobiellen Eiweissbausteinen ausgestattet sind. Erste Ergebnisse wurden bereits veröffentlicht und zeigen: Die hautfreundlichen Membranen aus Pflanzenmaterial töten Bakterien äusserst effizient ab. Fanden die Forschenden eine vielversprechende Lösung gegen antibiotikaresistente Erreger?
Andrea Six ¹
Dringen Keime in eine Wunde ein, können sie eine dauerhafte Infektion auslösen, die nicht abheilen mag. Die Infektion kann sich sogar im Körper ausbreiten und zu einer lebensgefährlichen Blutvergiftung (Sepsis) führen kann. Gerade bei komplexen Wunden stellt sich heute immer häufiger das Problem der Antibiotika-Resistenz, da Bakterien wie etwa Staphylokokken unempfindlich gegen die einstige Wunderwaffe der Medizin geworden sind. Forschende der Empa haben daher Cellulose-Membranen entwickelt, mit denen sich derartige Infektionen im Keim ersticken lassen. Das Team um Empa-Forscherin Katharina Maniura vom «Biointerfaces»-Labor in St. Gallen stellte hierzu feine Membranen aus pflanzlicher Cellulose mittels Electrospinning her. Die Cellulosefasern mit einem Durchmesser unter einem Mikrometer wurden zu einem zarten dreidimensionalen Gewebe in mehreren Schichten gesponnen. Besonders flexibel und gleichzeitig stabil wurden die Membranen, nachdem die Forschenden zusätzlich das Polymer Polyurethan mit eingesponnen hatten. Um einen antibakteriellen Effekt zu erzielen, entwarfen die Forschenden multifunktionale Eiweissbausteine – also Peptide –, die sich einerseits an die Cellulosefasern binden können und zudem eine antimikrobielle Aktivität aufweisen. Diese Peptide haben den Vorteil, dass sie einfacher herzustellen sind und stabiler bleiben als grössere Biomoleküle wie Proteine, die empfindlicher auf die chemischen Bedingungen in einer Wunde reagieren.
Staphylokokken im Rasterelektronenmikroskop.

Hautfreundliche Membranen
Behandelte man die Cellulose-Membranen mit einer derartigen Peptidlösung, sättigte sich das Faserngerüst mit den Eiweissbausteinen. In Zellkulturexperimenten wiesen die Forschenden daraufhin nach, dass die Peptid-haltigen Membranen für menschliche Hautzellen gut verträglich sind. Für Bakterien wie Staphylokokken, die häufig in schlechtheilenden Wunden zu finden sind, waren die Cellulose-Membranen hingegen ein Todesurteil. «In Bakterienkulturen wurden über 99.99 Prozent der Keime durch die Peptid-haltigen Membranen abgetötet», so Maniura. Künftig sollen die antimikrobiellen Membranen zudem mit weiteren Funktionen ausgestattet werden. «Die Peptide könnten beispielsweise mit Bindungsstellen funktionalisiert werden, die eine kontrollierte Abgabe von weiteren therapeutischen Wirkstoffen ermöglichen», so Maniura.
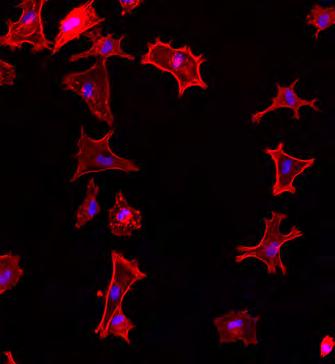
Yoga für Bindegewebszellen: In Gegenwart der multifunktionalen Eiweissbausteine einer Cellulose-Oberfläche heften sich menschliche Bindegewebszellen (Fibroblasten), wie erhofft, besser an und beginnen, sich gleichmässig zu verteilen.
Medienmitteilung Empa www.empa.ch










